SBC ToolBox在Small RNA-seq定量数据分析中的应用(上)
Small RNA-seq数据量那么少,又不需要重新比对定量,简简单单的做个差异分析、画个图(火山图、heatmap)、PCA、相关性计算、筛选下差异基因、预测下miRNA的靶基因,靶基因GO&KEGG注释,使用SBC ToolBox三下五除二,只要你手速快,结果就立马展现你的面前。
在开启使用SBC ToolBox针对Small RNA-seq定量数据之前,先来认识一下数据,对数据有充分认知才能更好的进行数据分析。
一 数据认知
i. 表达定量数据
a) 手里的数据可能是这个样子,一般都是.xlsx格式,如果不是请使用Excel或者WPS进行另存为.xlsx格式数据。如下图:
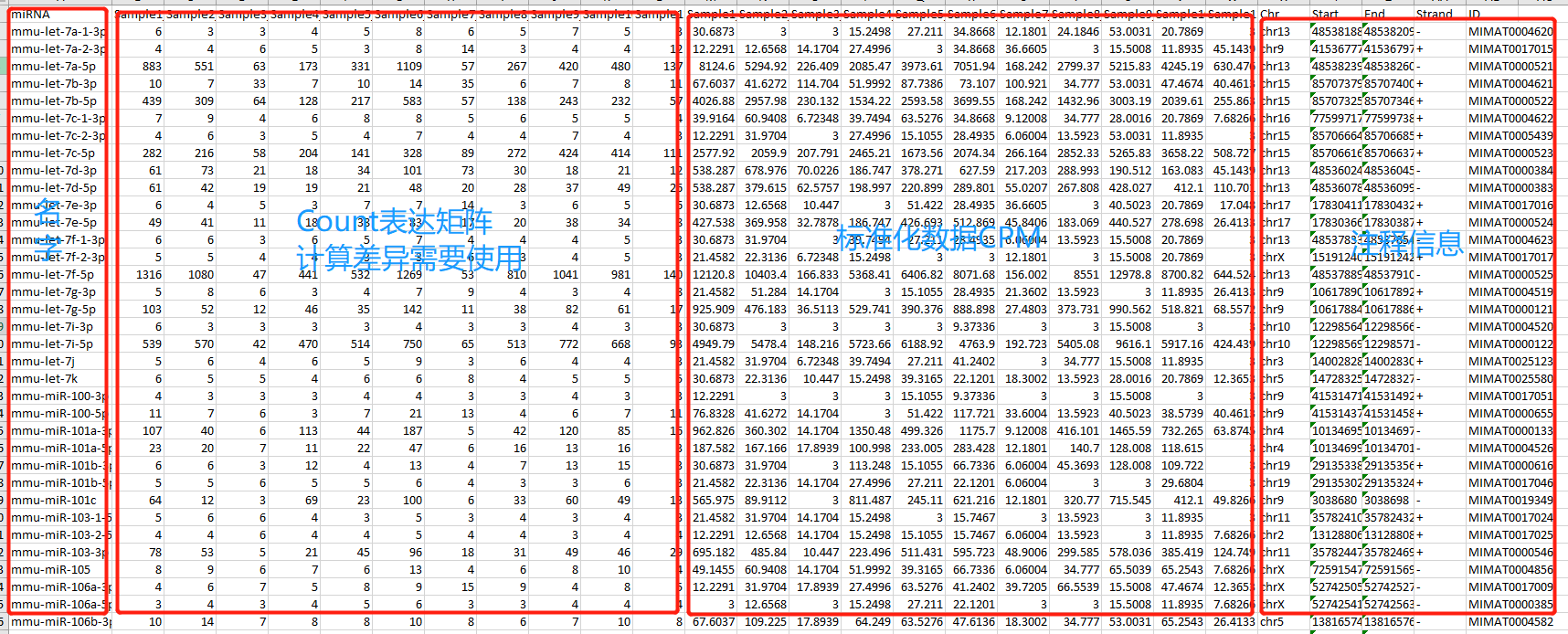
b) 定量结果可以拆成四部分。
1. 第一列:Small RNA名字,重要,计算差异基因名字;
2. Count表达矩阵列,常常说的定量结果,也是edgeR或者DESeq2等软件的输入数据;
3. CPM列,对Count标准化后的数据,一般用于PCA、层级聚类、相关性计算;
4. 注释信息:对Small RNA的位置或者其他信息注释。
ii. 样本分组数据
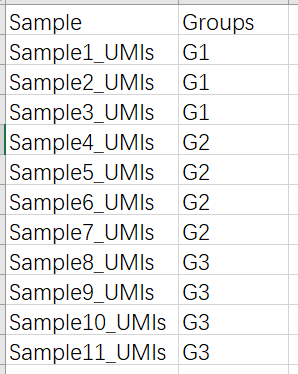
大多数实验都有生物学重复,也就是多个样本对应一个分组的情况,这里需要的文件格式是下图格式,第一列为样品信息,一定要与定量表格中的样品信息一样,差一点都不行,第二列就是样品对应的分组信息,这里只支持.xlsx格式文件,没有这个文件可以使用Excel或者WPS手搓(从定量表格中Ctrl+C & Ctrl+V样品信息,一定不会出问题,分组信息手动填写):
iii. 分组比较数据
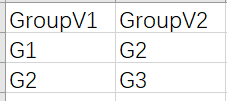
这就更简单了,一共就两列,第一列为Case组,第二列为Control组,这个要根据实际情况机型调整,注意:比较信息一定要与样品分组信息中的第二列匹配。这个文件没有,新建.xlsx直接手搓,你就会体验到手搓的快乐。
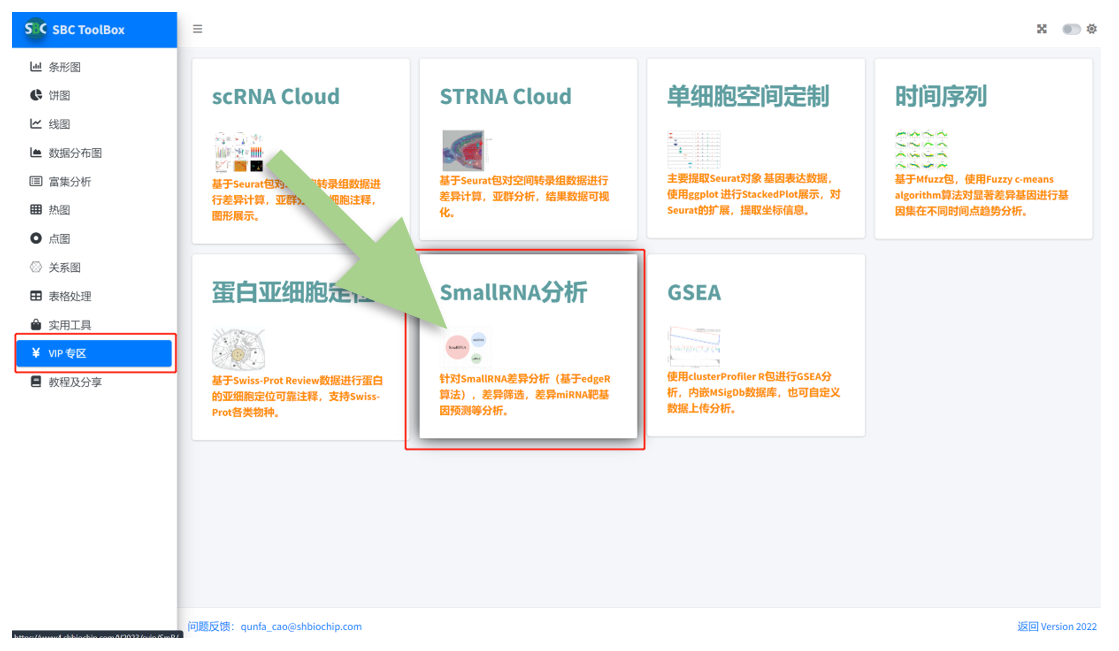
二 登录SBC ToolBox SmallRNA模块
i. 方法一
导航入口:https://www4.shbiochip.com/SBCToolBox/
ii. 方法二
一键直达:https://www4.shbiochip.com/V2023/svip/SmR/
iii. 登录验证
a) 登录界面
b) 进入分析界面:
三 SBC ToolBox差异分析
i. 进入数据上传&差异分析组件

a) 数据上传
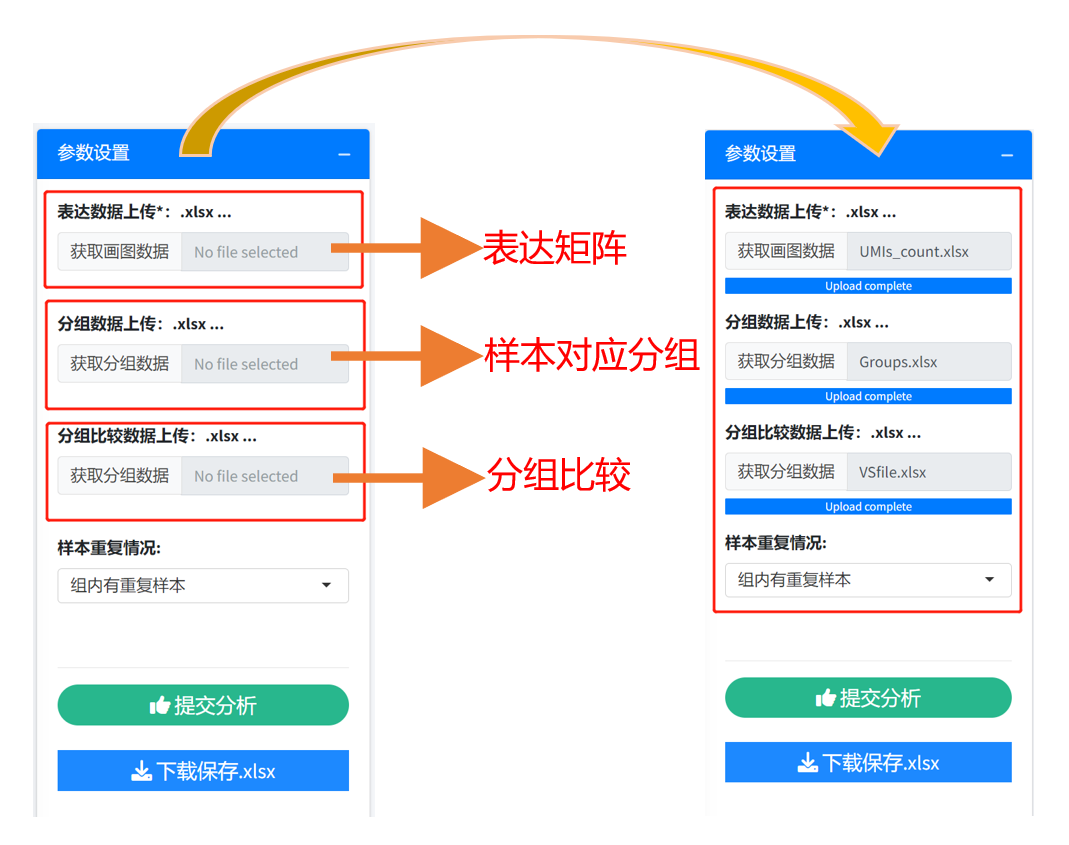
直接将准备好的数据文件拖拽或者点击上传接口,上传到云服务器上。
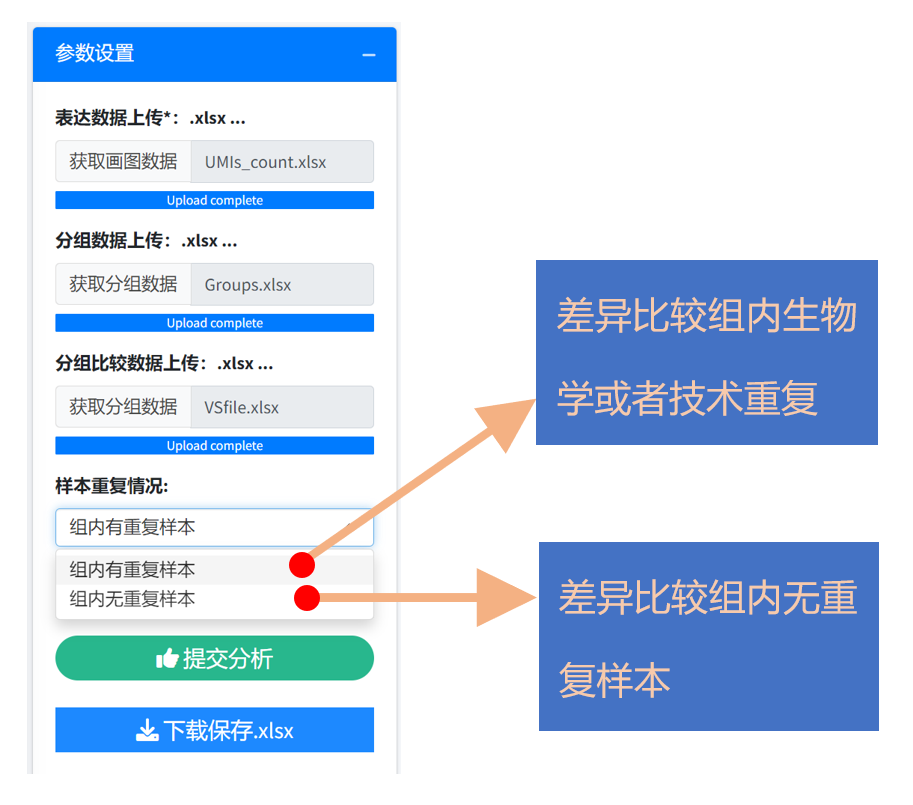
b) 注意:
c) 提交分析&结果预览
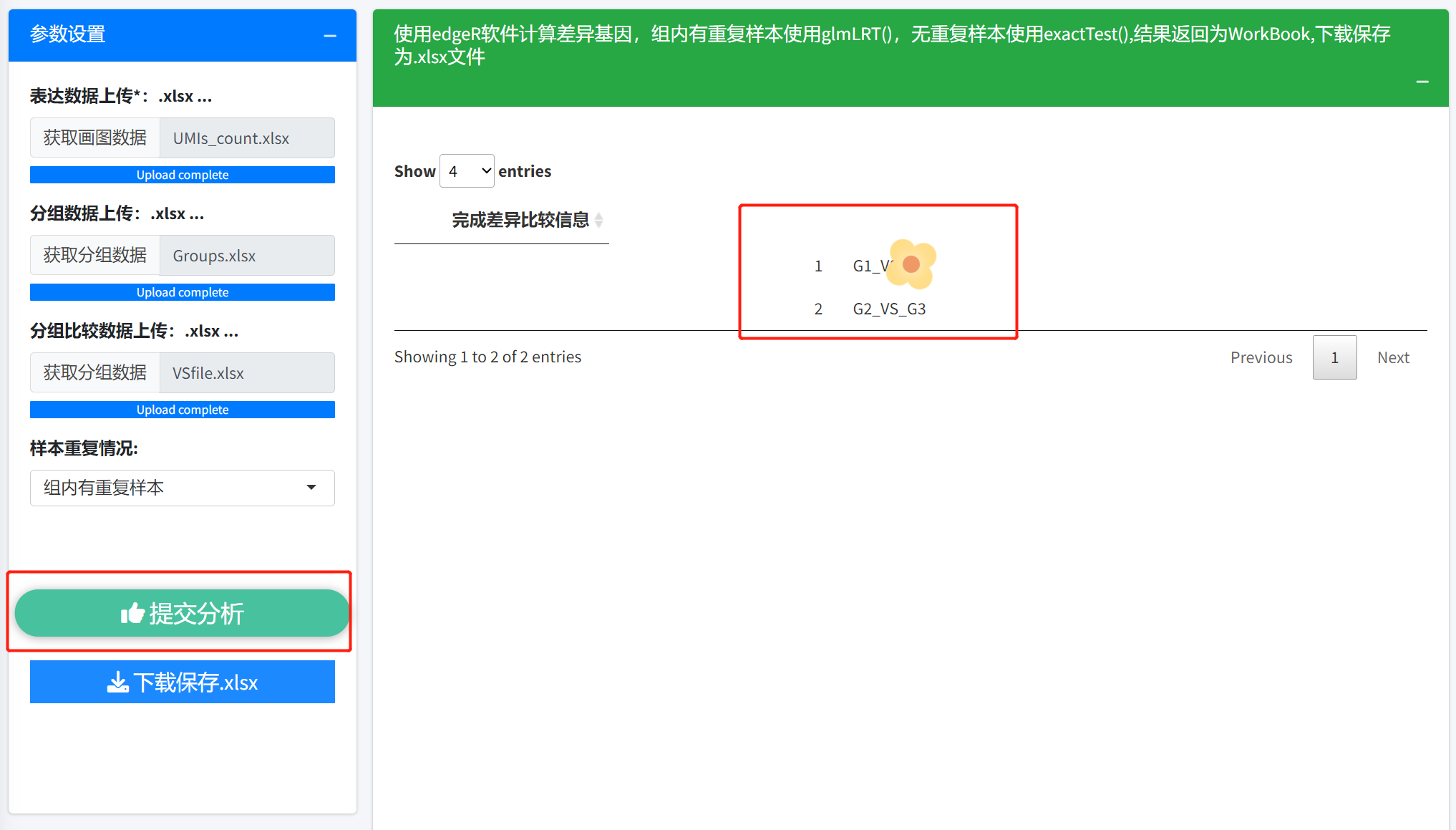
点击提交分析按钮,主界面会有一个小花花转动提示正在运行,分析完成后,小花花消失,主界面显示分组比较信息。整个过程不超过半分钟。
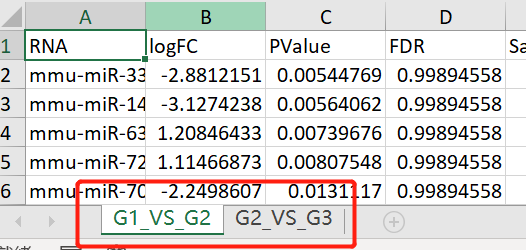
d) 完成分析后,直接点击 下载保存.xlsx 按钮,结果将会以.xlsx格式的workbook,不同的分组比较将会保存在不同的sheet中。
ii. 差异结果阈值筛选组件

默认情况下保存的差异结果为不筛选的,可以使用差异结果阈值筛选组件根据pvalue和差异倍数(FC)进行阈值筛选。
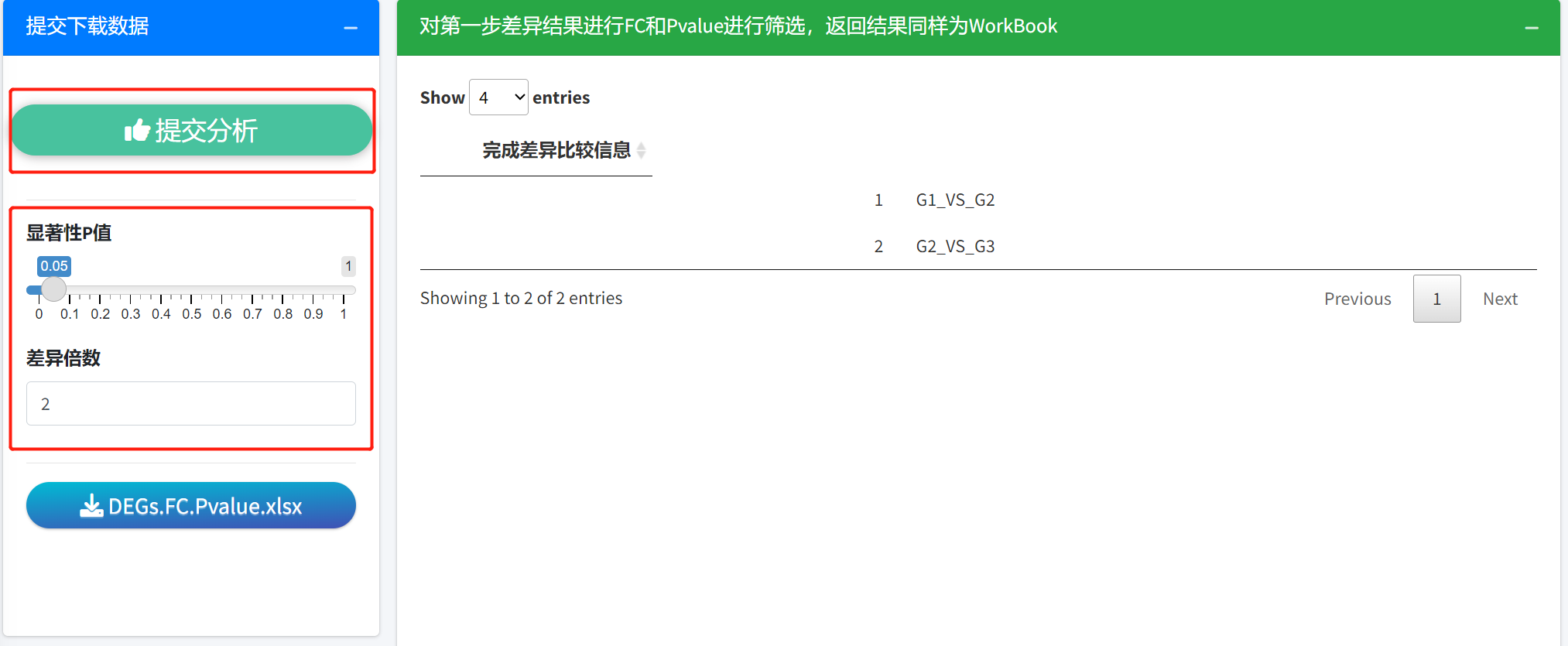
这里只需要设置简单的设置pvalue 以及 差异倍数阈值,App会自动筛选出目标阈值的差异结果,提交分析,下载保存结果即可完成分析,无需等待分分钟完成分析任务。
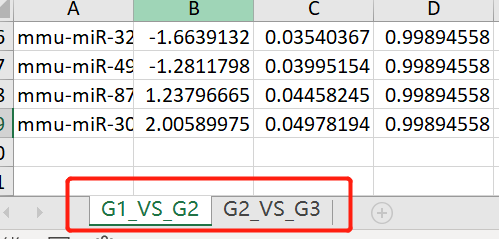
直接点击DEGs.FC.Pvalue.xlsx 按钮就可以下载保存筛选结果。结果仍旧为workbook,不同的sheet对应不同的分组比较阈值筛选结果。
iii. 靶基因预测组件

a) 差异靶基因预测
1. 对接差异分析结果
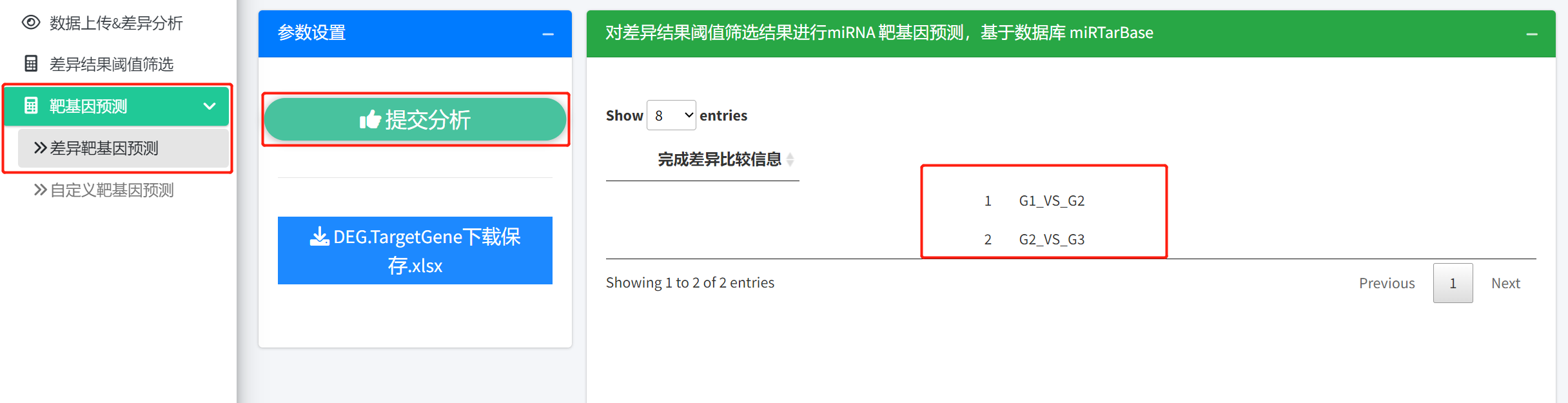
这一部分向上承接筛选阈值后的差异miRNA的预测。结果仍旧为workbook,直接点击下载保存结果即可保存结果到本地。
b) 自定义靶基因预测
1. 自定义输入miRNA筛选靶基因
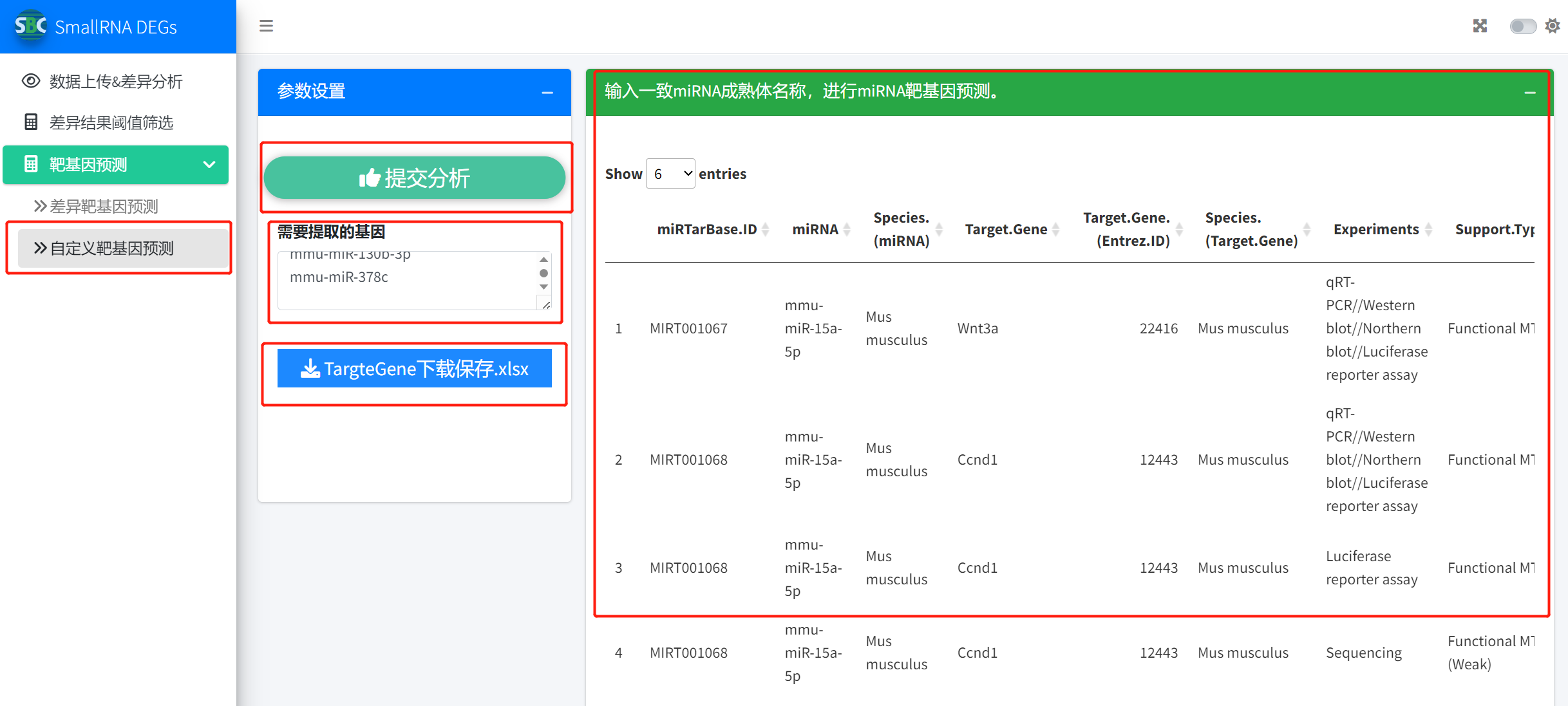
主要针对目标miRNA进行靶基因预测,这里只需要将一列目标miRNA复制粘贴至目标区域,提交分析即可完成miRNA靶基因查询,主界面会预览结果,点击 TargetGene下载保存.xlsx 按钮即可保存结果。
完成以上操作步骤,即可完成miRNA的差异分析,如果你的数据是其他类型的SmallRNA,也可以完成差异分析和筛选,无法进行靶基因预测。
以上就是本期Small RNA-seq定量数据分析上半部分的介绍,下期将紧接本期内容,为您带来「检验样本重复性模块」、「差异数据展示模块」和「靶基因的GO&KEGG注释」等下半部分的详细介绍,敬请期待。
SBC ToolBox共有十二大类103种绘图工具,注册后即可免费绘制各种自定义图片,快来看看吧。





