植物组学时代的DNA甲基化研究在群体表观遗传学中的应用
DNA甲基化在许多生物过程中发挥着重要作用。得益于数十年来对DNA甲基化突变体的研究,如今对DNA甲基化的建立和维持机制有了很好的理解,尤其在拟南芥(Arabidopsis thaliana)的Col-0品系中的研究。最近的研究利用自然品系甲基化组进行的全基因组关联研究(GWASs)从群体水平上揭示了DNA甲基化变化的复杂而独特的遗传基础。亚硫酸盐处理后的测序是量化DNA甲基化的杰出方法,此外最近开发的无亚硫酸盐检测甲基化方法,以及同时检测遗传和表观遗传信息的具有成本效益的方法。本文还讨论了特定拟南芥品系中DNA甲基化的可塑性,DNA甲基化在植物适应环境中的作用,以及自然群体中DNA甲基化变化的遗传决定因素。最近开发的技术和知识将极大地促进未来在群体表观基因组学领域的研究。
DNA甲基化全基因组检测技术
基于亚硫酸盐的二代测序
DNA甲基化研究的第一步是在特定位点或全基因组范围内检测和定量甲基化水平。为了实现这一目标,已经开发了数十种方法,在这些方法中,基于亚硫酸盐的方法使用最为广泛。全基因组亚硫酸盐测序(WGBS)已成为在单碱基分辨率下定量检测DNA甲基化的金标准。亚硫酸盐处理DNA时,会将未甲基化的胞嘧啶转化为尿嘧啶,而甲基化的胞嘧啶则保持不变。经过PCR扩增后,未甲基化的胞嘧啶在测序时会呈现为胸腺嘧啶。将WGBS数据与参考序列比对后,根据特定位点上胸腺嘧啶比例鉴定每个胞嘧啶的甲基化水平。为准确定量甲基化水平,WGBS覆盖度要求通常比基于单核苷酸多态性鉴定的全基因组测序(WGS)更高。可能会限制WGBS在群体水平上对大基因组物种的应用,如玉米(Zea mays)(2.1 Gb)和小麦(Triticum aestivum,14.5Gb)。为克服这一限制开发出多种替代策略来捕获目标基因组区域的DNA甲基化,如简化基因组亚硫酸盐测序(RRBS)、甲基化DNA免疫沉淀测序(MeDIP-seq)、全基因组DNA甲基化芯片以及修饰胞嘧啶转化捕获方法。
不基于亚硫酸盐的甲基化测序
基于亚硫酸盐测序方法的局限在于文库制备和甲基化比对中的固有偏差,这主要是由于在胞嘧啶转化反应过程中亚硫酸盐处理条件严格,难以将短读长唯一比对到包含重复DNA序列的基因组区域。偏差可能是未甲基化胞嘧啶富集的基因组区域更快降解、胞嘧啶转化不完全以及由碱基含量偏倚引起的PCR偏差。近年来为减少这种偏差,开发许多无亚硫酸盐策略,如通过第三代测序或基于酶促反应的测序技术。
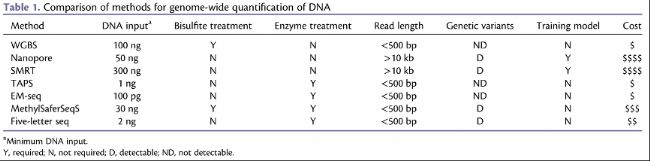
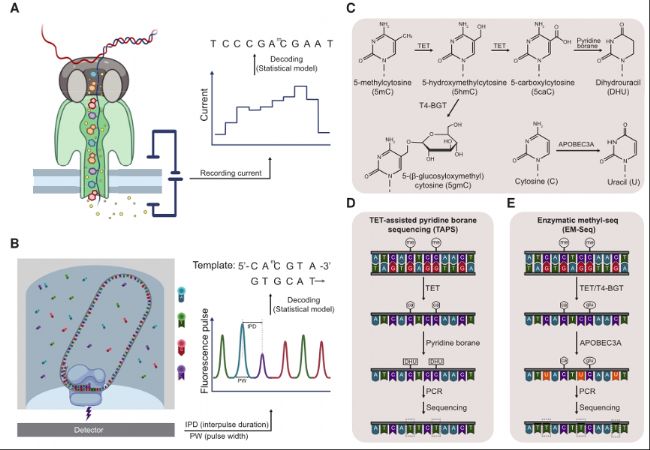
图1:用于定量检测DNA甲基化的无亚硫酸盐方法。
A) Nanopore长读长测序。
B) SMRT长读长测序。
C-E) 两种基于酶促的测序方法。
同时检测遗传和表观遗传信息
MethylSaferSeqS和5-letter-seq能够同时检测遗传信息和甲基化状态。与亚硫酸盐文库制备相比,MethylSaferSeqS在PCR后将分离原始DNA模板和扩增产物,分别用作后续WGBS和WGS的input文库。而5-letter-seq在单个文库中保留了遗传和表观遗传信息,将片段化DNA的两端连接到含有尿嘧啶残基的发夹接头上,随后使用尿嘧啶特异性切除试剂处理,将两条DNA链分开;合成cDNA链后,得到的扩增子形成发夹结构,一条链包含原始的表观遗传信息,互补链保留遗传信息。在与测序接头连接后,甲基化胞嘧啶通过TET和BGT被APOBEC3A保护免受氧化,而未甲基化胞嘧啶则通过APOBEC3A和UvrD解旋酶转化为尿嘧啶。甲基化胞嘧啶保持不变,未甲基化胞嘧啶在最终的测序reads片段被转化为胸腺嘧啶。根据解码规则,可以从每个发夹的测序reads片段中区分甲基化和未甲基化胞嘧啶。5-letter-seq可以在单个文库中确定遗传序列和DNA甲基化水平,大幅降低群体中甲基化研究成本。这些方法在群体表观基因组学领域具有巨大的应用潜力,该领域需要关于基因突变和甲基化模式信息,特别对于大基因组物种。
单细胞DNA甲基化分析
在活细胞中,DNA胞嘧啶应该处于两种状态之一(即甲基化或未甲基化)。然而,由于细胞异质性和组织及个体样本混合,甲基化水平经常在未甲基化到完全甲基化之间定量变化。在过去十年中,已经开发了十多种方法,用于从单一细胞中分析哺乳动物的DNA甲基化,包括单细胞简化亚硫酸盐测序(scRRBS)、单细胞亚硫酸盐测序(scBS-seq)、单细胞CpG岛甲基化测序(scCGI-seq);Smart-RRBS用于单个细胞中的DNA甲基化和转录组分析;单细胞多组学测序技术(scNOMeRe-seq)用于从同一个单细胞中同时进行染色质可及性、DNA甲基化和RNA表达的全基因组分析。与哺乳动物中这些技术的快速发展相比,植物中单细胞DNA甲基化的分析进展要慢得多。虽然植物中的大多数单细胞研究都集中在转录组上,但有研究使用亚硫酸盐转化的随机整合片段测序(BRIF-seq)揭示了玉米中4个四分体的16个微孢子的单细胞DNA甲基化组。WGBS结合荧光激活细胞分选,已经允许揭示特定细胞类型的DNA甲基化组,包括6种不同的根分生组织细胞群、精子和营养细胞、茎和非茎的芽分生组织细胞。这些研究揭示了植物细胞的异质性和不同细胞类型中DNA甲基化的动态变化。
DNA甲基化可塑性
拟南芥品系Col-0的DNA甲基化变化
在个体水平上,DNA甲基化动态变化,并且可以在来自同一祖先的植物代际间变化,也可以在不同实验室中同一植物的不同组织以及来自相同组织的细胞之间变化。不同实验室中Col-0基因组DNA甲基化水平差异在CG中高达10%、在非CG甲基化区域达到7%。DNA甲基化模式表现出与上下文(CG、CHG、CHH)背景差异,在发育的各个阶段观察到CG甲基化稳定、CHG甲基化增加、CHH甲基化减少。且与发育相关的大多数甲基化变化发生在着丝粒转座元件(TEs)中。
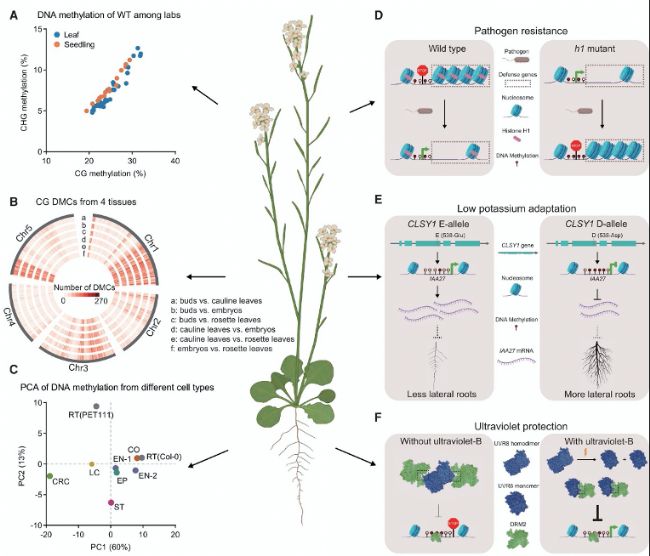
图 2. DNA甲基化可塑性及其对植物适应环境中的作用。
A. 来自不同实验室的50个高质量WGBS数据集的野生型(WT)拟南芥Col-0的全基因组平均CG和CHG甲基化水平。
B. 通过比较4种组织的DNA甲基化数据鉴定的CG DMCs分布。
C. 来自9种根细胞类型的100 kb窗口中CG、CHG和CHH甲基化水平的主成分分析(PCA)。RT,根尖;EP,表皮;CO,皮层;EN,内皮层;ST,髓;CRC,柱状根帽;LC,下柱状。
D. DNA甲基化对植物对免疫启动的敏感性。
E. IAA27的DNA甲基化变化与CLSY1的基因突变相结合,影响侧根发育并赋予其对低钾环境的适应。"T"形实线表示DNA甲基化增加抑制IAA27表达。"T"形虚线表示IAA27表达负调控侧根发育,较粗线条表示更高表达抑制作用更强。
F. 在暴露于UVB后,UVR8-DRM2模块诱导的DNA甲基化减少激活了下游基因/TEs。虚线框表示UVR8和DRM2之间的物理作用位点。"T"形实线表示抑制了DRM2介导的DNA甲基化,较粗线条具有更强抑制作用。
相比之下,组织(如根和茎)之间的甲基化模式差异呈动态变化,其中在常染色质区域的CG甲基化和着丝粒区域的非CG甲基化变化具有显著差异。这种表观基因组的可塑性主要归因于自发的表观遗传变化。与基因突变相比,表观遗传变化的发生率高得多,且在拟南芥基因组中已经鉴定出表观遗传突变热点区域。尽管这些区域只占基因组中CG位点的约12%,但在这些热点区域观察到约63%的表观遗传突变事件。甲基化模式差异的另一个重要因子是甲基化机制的组织/细胞特异性表达。已知有十个基因参与表观遗传调控,包括DNA甲基化减少1(DDM1)、ARGONAUTE 9(AGO9)和SU(VAR)3-9同源物4(SUVH4),这些基因在干细胞中表达上调。CLASSY家族基因表现出组织特异性表达模式;这些蛋白以组织特异性方式调控24-nt siRNA产生和DNA甲基化。活性DNA去甲基化是组织间差异性甲基化的另一因素,拟南芥DNA去甲基化由4个DEMETER家族成员—DME、DML2、DML3和ROS1调控,野生型中鉴定出的许多组织特异性甲基化变化在这4个基因的四重突变体中都不存在。
响应环境的DNA甲基化变化
越来越多的研究表明,DNA甲基化作为一种可遗传的胞嘧啶修饰,能够响应并适应各种生物和非生物胁迫。本文重点描述DNA甲基化在植物适应生物和非生物胁迫中重要作用的最新研究。
虽然基因突变赋予了植物长期适应性,但DNA甲基化可以快速响应环境挑战并迅速提高适应度。Sheikh等人(2023年)发现了接头组蛋白H1和DNA甲基化在植物防御中的重要作用。由于植物防御基因表达变化,一个含有3种组蛋白H1变体(h1.1、h1.2、h1.3)的拟南芥三重突变体表现出对植物病原体感染抗性增强。在病原体感染之前使用22-氨基酸鞭毛蛋白(flg22)进行预处理的启动过程增强了野生型植物对随后病原体感染抗性。然而h1突变体对启动不敏感。flg22处理导致h1防御基因启动子中的DNA甲基化增加,从而导致其抑制。这项研究表明,DNA甲基化可以快速响应病原体感染,并通过调控防御基因表达影响植物防御。
DNA甲基化也可以在长期进化过程中增强植物的适应性,无论是依赖或独立于基因突变。Shahzad等人(2020年)最近研究表明,DNA甲基化变化与基因突变相结合,允许植物响应钾缺乏。对土壤中营养元素的感知和反应对植物至关重要,必需营养素钾的缺乏可能导致根系发育不良。不同的拟南芥品系已经进化出2种策略,通过增加主根或侧根的生长来克服低钾胁迫。Shahzad等人(2020年)使用全基因组关联研究(GWAS)鉴定出CLSY1作为低钾条件下侧根发育的调控因子。低钾抑制了吲哚-3-乙酸诱导27(IAA27)的降解,IAA27通过生长素信号通路负向调控根分枝。同时CLSY1可以通过DNA甲基化沉默IAA27。在CLSY1的538位点上,天冬氨酸到谷氨酸变化与自然拟南芥品系中低钾条件下侧根发育显著相关。携带编码天冬氨酸的CLSY1等位基因品系在IAA27启动子上的DNA甲基化水平显著增加,与携带编码谷氨酸的等位基因品系相比,IAA27表达水平显著下调。这些发现表明,DNA甲基化可以协调遗传变化,并促进侧根发育,使植物能够克服具有挑战性的环境。
尽管DNA甲基化已被认为与植物适应有关,但证据大多来自显示DNA甲基化变化与环境变化相关的研究。Jiang等人(2021年)提供了直接证据来说明紫外线照射如何抑制DNA甲基化。拟南芥中的DNA甲基化通过UVB光响应,主要通过UV RESISTANCE LOCUS 8(UVR8,一种UVB光受体)和DRM2(一种新的DNA甲基转移酶)之间的物理互作实现。UVB照射通过UVR8依赖性通路诱导全基因组DNA低甲基化并解除转座元件(TEs)抑制。这种UVR8-DRM2介导的TEs重新激活机制可能直接或间接调控参与植物抵御紫外线照射的关键基因表达。
自然群体中DNA甲基化变化的遗传基础
对特定基因组位点以及不同拟南芥品系的全基因组甲基化组的研究显示,自然品系间的DNA甲基化存在显著差异。与在个别品系中已充分描述的DNA甲基化建立和维持的调控因子相比,自然品系间DNA甲基化变化的遗传和分子机制了解甚少。GWAS已经鉴定出此过程的多个影响因子,其中CMT2因全基因组非CG甲基化水平、差异甲基化区域(DMRs)和CMT2靶向的转座元件(TEs)被多个GWAS定位。这与CMT2作为负责维持CHH甲基化的DNA甲基转移酶功能以及CHH和CHG甲基化之间的强相关性相吻合。另一个TEs上CHH甲基化的主要决定因子是NRPE1,它编码RNA聚合酶V的最大亚基,是RNA介导DNA甲基化(RdDM)通路的关键组分。NRPE1在调控TEs移动性方面的作用也通过GWAS揭示。
在一项GWAS中,miR823A通常与CHG DMRs相关。将CHH甲基化作为协变量后,另一项RdDM/CMT2靶向TEs的CHG甲基化的GWAS鉴定出CMT3和miR823A。CMT3编码负责维持CHG甲基化的DNA甲基转移酶,而miR823A编码的microRNA823A预计靶向CMT3。这些发现表明miR823A-CMT3模块介导的CHG甲基化调控可能,但还需要进一步验证。
包括AGO9和AGO1在内的ARGONAUTE基因被鉴定为自然群体中RdDM靶向TEs的CHH甲基化调控因子。这些基因在这些品系中的功能可能与Col-0中AGO9功能不同,因为ago9突变体在RdDM靶向的低甲基化区域(hypo-DMR)的甲基化没有变化。这表明在Col-0中没有参与RdDM通路的AGO蛋白可能在其他品系中参与DNA甲基化。
MULTICOPY SUPPRESSOR OF IRA1(MSI1)编码Polycomb抑制复合体2(PRC2)的一个亚基,该亚基催化组蛋白3赖氨酸27位点的三甲基化(H3K27me3)。在两项最近的GWAS中,MSI1与44个DMR区域的CHG甲基化以及RdDM和CMT2靶向的TEs相关。在Col-0背景下,H3K27me3与DNA甲基化无关,与CMT3对CHG甲基化所必需的H3K9me2不同。这些发现表明在其他品系中DNA甲基化和H3K27me3之间可能存在相关性,这有待进一步表征。
尽管没有被重复鉴定,JUMONJI26(JMJ26)—BONSAI METHYLATION 1(IBM1)的同源物基因突变被报告与使用CHH作为协变量的条件GWAS中RdDM靶向TEs的CHG甲基化相关联。敲除JMJ26导致RdDM靶向TEs的CHG甲基化增加。鉴定JMJ26的关键是在CHG甲基化的GWAS时将CHH甲基化作为协变量的条件分析。这一发现突出了GWAS在群体表观遗传学研究中的创新潜力,包括研究方法或群体构建。
除了转录调控因子外,大量的基因突变,无论是邻近还是远端,已被证明与靶位点的DNA甲基化变化相关联。尽管如此,关于这些关联背后的因果基因突变知之甚少,因为还没有验证这些关联背后的遗传变化。一些研究表明,诸如TE插入和结构变异(SVs)的基因突变可以影响附近位点的DNA甲基化。Ahmed等人(2011年)发现,没有匹配24-nt siRNA的数百个TEs通过邻近密集甲基化的TEs甲基化扩散获得DNA甲基化。另一项研究发现,在211个拟南芥品系中,近一半的新TE插入位点在带有TE插入的品系中高度甲基化并扩散到邻近区域。直接证据来自CEN180重复序列在常染色质靶位点的从头沉积,从而在ibm1突变体中诱导局部DNA甲基化的建立和扩散。在拟南芥1001个甲基化组中,大部分SVs(22%至50%)的甲基化存在差异,表明了SV对邻近序列DNA甲基化的影响。SV如何影响DNA甲基化的最好例子是拟南芥中的PHOSPHORIBOSYLANTHRANILATE ISOMERASE(PAI)基因家族(PAI1至PAI4),一些品系包含未甲基化的PAI基因,如Col,而其他品系包含甲基化的PAI基因,如WS。Col包含3个不连锁的PAI基因(PAI1、PAI2和PAI3),而WS包含4个PAI基因,具有PAI1至PAI4的倒置重复。这个倒置重复导致所有4个PAI基因甲基化。
易小结
最近的全基因组关联研究(GWASs)为理解转座元件(TEs)非CG甲基化的自然变异的遗传基础提供了重要信息。然而目前对CG甲基化的遗传基础以及其他基因组特征(如启动子区域和蛋白质编码基因)的DNA甲基化了解仍然知之甚少。高通量检测和验证表观遗传变化的功能,以及将这些变化在作物育种项目中的应用,是未来重要的研究工作。在群体水平上的包含多组学数据集的全基因组染色质图谱的可用性,无疑将加深对表观遗传变化的理解。结合大数据分析与表观遗传编辑技术,用于挖掘和应用表观遗传变化,将极大增强作物育种项目。
未解决的问题
Liu J, Zhong X. Population epigenetics: DNA methylation in the plant omics era. Plant Physiol. 2024 Feb 16. pii: 7609601. doi: 10.1093/plphys/kiae089. PubMed PMID: 38366882.
DNA甲基化全基因组检测技术
基于亚硫酸盐的二代测序
DNA甲基化研究的第一步是在特定位点或全基因组范围内检测和定量甲基化水平。为了实现这一目标,已经开发了数十种方法,在这些方法中,基于亚硫酸盐的方法使用最为广泛。全基因组亚硫酸盐测序(WGBS)已成为在单碱基分辨率下定量检测DNA甲基化的金标准。亚硫酸盐处理DNA时,会将未甲基化的胞嘧啶转化为尿嘧啶,而甲基化的胞嘧啶则保持不变。经过PCR扩增后,未甲基化的胞嘧啶在测序时会呈现为胸腺嘧啶。将WGBS数据与参考序列比对后,根据特定位点上胸腺嘧啶比例鉴定每个胞嘧啶的甲基化水平。为准确定量甲基化水平,WGBS覆盖度要求通常比基于单核苷酸多态性鉴定的全基因组测序(WGS)更高。可能会限制WGBS在群体水平上对大基因组物种的应用,如玉米(Zea mays)(2.1 Gb)和小麦(Triticum aestivum,14.5Gb)。为克服这一限制开发出多种替代策略来捕获目标基因组区域的DNA甲基化,如简化基因组亚硫酸盐测序(RRBS)、甲基化DNA免疫沉淀测序(MeDIP-seq)、全基因组DNA甲基化芯片以及修饰胞嘧啶转化捕获方法。
不基于亚硫酸盐的甲基化测序
基于亚硫酸盐测序方法的局限在于文库制备和甲基化比对中的固有偏差,这主要是由于在胞嘧啶转化反应过程中亚硫酸盐处理条件严格,难以将短读长唯一比对到包含重复DNA序列的基因组区域。偏差可能是未甲基化胞嘧啶富集的基因组区域更快降解、胞嘧啶转化不完全以及由碱基含量偏倚引起的PCR偏差。近年来为减少这种偏差,开发许多无亚硫酸盐策略,如通过第三代测序或基于酶促反应的测序技术。

表1:全基因组DNA甲基化定量方法比较

图1:用于定量检测DNA甲基化的无亚硫酸盐方法。
B) SMRT长读长测序。
C-E) 两种基于酶促的测序方法。
同时检测遗传和表观遗传信息
MethylSaferSeqS和5-letter-seq能够同时检测遗传信息和甲基化状态。与亚硫酸盐文库制备相比,MethylSaferSeqS在PCR后将分离原始DNA模板和扩增产物,分别用作后续WGBS和WGS的input文库。而5-letter-seq在单个文库中保留了遗传和表观遗传信息,将片段化DNA的两端连接到含有尿嘧啶残基的发夹接头上,随后使用尿嘧啶特异性切除试剂处理,将两条DNA链分开;合成cDNA链后,得到的扩增子形成发夹结构,一条链包含原始的表观遗传信息,互补链保留遗传信息。在与测序接头连接后,甲基化胞嘧啶通过TET和BGT被APOBEC3A保护免受氧化,而未甲基化胞嘧啶则通过APOBEC3A和UvrD解旋酶转化为尿嘧啶。甲基化胞嘧啶保持不变,未甲基化胞嘧啶在最终的测序reads片段被转化为胸腺嘧啶。根据解码规则,可以从每个发夹的测序reads片段中区分甲基化和未甲基化胞嘧啶。5-letter-seq可以在单个文库中确定遗传序列和DNA甲基化水平,大幅降低群体中甲基化研究成本。这些方法在群体表观基因组学领域具有巨大的应用潜力,该领域需要关于基因突变和甲基化模式信息,特别对于大基因组物种。
单细胞DNA甲基化分析
在活细胞中,DNA胞嘧啶应该处于两种状态之一(即甲基化或未甲基化)。然而,由于细胞异质性和组织及个体样本混合,甲基化水平经常在未甲基化到完全甲基化之间定量变化。在过去十年中,已经开发了十多种方法,用于从单一细胞中分析哺乳动物的DNA甲基化,包括单细胞简化亚硫酸盐测序(scRRBS)、单细胞亚硫酸盐测序(scBS-seq)、单细胞CpG岛甲基化测序(scCGI-seq);Smart-RRBS用于单个细胞中的DNA甲基化和转录组分析;单细胞多组学测序技术(scNOMeRe-seq)用于从同一个单细胞中同时进行染色质可及性、DNA甲基化和RNA表达的全基因组分析。与哺乳动物中这些技术的快速发展相比,植物中单细胞DNA甲基化的分析进展要慢得多。虽然植物中的大多数单细胞研究都集中在转录组上,但有研究使用亚硫酸盐转化的随机整合片段测序(BRIF-seq)揭示了玉米中4个四分体的16个微孢子的单细胞DNA甲基化组。WGBS结合荧光激活细胞分选,已经允许揭示特定细胞类型的DNA甲基化组,包括6种不同的根分生组织细胞群、精子和营养细胞、茎和非茎的芽分生组织细胞。这些研究揭示了植物细胞的异质性和不同细胞类型中DNA甲基化的动态变化。
DNA甲基化可塑性
拟南芥品系Col-0的DNA甲基化变化
在个体水平上,DNA甲基化动态变化,并且可以在来自同一祖先的植物代际间变化,也可以在不同实验室中同一植物的不同组织以及来自相同组织的细胞之间变化。不同实验室中Col-0基因组DNA甲基化水平差异在CG中高达10%、在非CG甲基化区域达到7%。DNA甲基化模式表现出与上下文(CG、CHG、CHH)背景差异,在发育的各个阶段观察到CG甲基化稳定、CHG甲基化增加、CHH甲基化减少。且与发育相关的大多数甲基化变化发生在着丝粒转座元件(TEs)中。

图 2. DNA甲基化可塑性及其对植物适应环境中的作用。
B. 通过比较4种组织的DNA甲基化数据鉴定的CG DMCs分布。
C. 来自9种根细胞类型的100 kb窗口中CG、CHG和CHH甲基化水平的主成分分析(PCA)。RT,根尖;EP,表皮;CO,皮层;EN,内皮层;ST,髓;CRC,柱状根帽;LC,下柱状。
D. DNA甲基化对植物对免疫启动的敏感性。
E. IAA27的DNA甲基化变化与CLSY1的基因突变相结合,影响侧根发育并赋予其对低钾环境的适应。"T"形实线表示DNA甲基化增加抑制IAA27表达。"T"形虚线表示IAA27表达负调控侧根发育,较粗线条表示更高表达抑制作用更强。
F. 在暴露于UVB后,UVR8-DRM2模块诱导的DNA甲基化减少激活了下游基因/TEs。虚线框表示UVR8和DRM2之间的物理作用位点。"T"形实线表示抑制了DRM2介导的DNA甲基化,较粗线条具有更强抑制作用。
相比之下,组织(如根和茎)之间的甲基化模式差异呈动态变化,其中在常染色质区域的CG甲基化和着丝粒区域的非CG甲基化变化具有显著差异。这种表观基因组的可塑性主要归因于自发的表观遗传变化。与基因突变相比,表观遗传变化的发生率高得多,且在拟南芥基因组中已经鉴定出表观遗传突变热点区域。尽管这些区域只占基因组中CG位点的约12%,但在这些热点区域观察到约63%的表观遗传突变事件。甲基化模式差异的另一个重要因子是甲基化机制的组织/细胞特异性表达。已知有十个基因参与表观遗传调控,包括DNA甲基化减少1(DDM1)、ARGONAUTE 9(AGO9)和SU(VAR)3-9同源物4(SUVH4),这些基因在干细胞中表达上调。CLASSY家族基因表现出组织特异性表达模式;这些蛋白以组织特异性方式调控24-nt siRNA产生和DNA甲基化。活性DNA去甲基化是组织间差异性甲基化的另一因素,拟南芥DNA去甲基化由4个DEMETER家族成员—DME、DML2、DML3和ROS1调控,野生型中鉴定出的许多组织特异性甲基化变化在这4个基因的四重突变体中都不存在。
响应环境的DNA甲基化变化
越来越多的研究表明,DNA甲基化作为一种可遗传的胞嘧啶修饰,能够响应并适应各种生物和非生物胁迫。本文重点描述DNA甲基化在植物适应生物和非生物胁迫中重要作用的最新研究。
虽然基因突变赋予了植物长期适应性,但DNA甲基化可以快速响应环境挑战并迅速提高适应度。Sheikh等人(2023年)发现了接头组蛋白H1和DNA甲基化在植物防御中的重要作用。由于植物防御基因表达变化,一个含有3种组蛋白H1变体(h1.1、h1.2、h1.3)的拟南芥三重突变体表现出对植物病原体感染抗性增强。在病原体感染之前使用22-氨基酸鞭毛蛋白(flg22)进行预处理的启动过程增强了野生型植物对随后病原体感染抗性。然而h1突变体对启动不敏感。flg22处理导致h1防御基因启动子中的DNA甲基化增加,从而导致其抑制。这项研究表明,DNA甲基化可以快速响应病原体感染,并通过调控防御基因表达影响植物防御。
DNA甲基化也可以在长期进化过程中增强植物的适应性,无论是依赖或独立于基因突变。Shahzad等人(2020年)最近研究表明,DNA甲基化变化与基因突变相结合,允许植物响应钾缺乏。对土壤中营养元素的感知和反应对植物至关重要,必需营养素钾的缺乏可能导致根系发育不良。不同的拟南芥品系已经进化出2种策略,通过增加主根或侧根的生长来克服低钾胁迫。Shahzad等人(2020年)使用全基因组关联研究(GWAS)鉴定出CLSY1作为低钾条件下侧根发育的调控因子。低钾抑制了吲哚-3-乙酸诱导27(IAA27)的降解,IAA27通过生长素信号通路负向调控根分枝。同时CLSY1可以通过DNA甲基化沉默IAA27。在CLSY1的538位点上,天冬氨酸到谷氨酸变化与自然拟南芥品系中低钾条件下侧根发育显著相关。携带编码天冬氨酸的CLSY1等位基因品系在IAA27启动子上的DNA甲基化水平显著增加,与携带编码谷氨酸的等位基因品系相比,IAA27表达水平显著下调。这些发现表明,DNA甲基化可以协调遗传变化,并促进侧根发育,使植物能够克服具有挑战性的环境。
尽管DNA甲基化已被认为与植物适应有关,但证据大多来自显示DNA甲基化变化与环境变化相关的研究。Jiang等人(2021年)提供了直接证据来说明紫外线照射如何抑制DNA甲基化。拟南芥中的DNA甲基化通过UVB光响应,主要通过UV RESISTANCE LOCUS 8(UVR8,一种UVB光受体)和DRM2(一种新的DNA甲基转移酶)之间的物理互作实现。UVB照射通过UVR8依赖性通路诱导全基因组DNA低甲基化并解除转座元件(TEs)抑制。这种UVR8-DRM2介导的TEs重新激活机制可能直接或间接调控参与植物抵御紫外线照射的关键基因表达。
自然群体中DNA甲基化变化的遗传基础
对特定基因组位点以及不同拟南芥品系的全基因组甲基化组的研究显示,自然品系间的DNA甲基化存在显著差异。与在个别品系中已充分描述的DNA甲基化建立和维持的调控因子相比,自然品系间DNA甲基化变化的遗传和分子机制了解甚少。GWAS已经鉴定出此过程的多个影响因子,其中CMT2因全基因组非CG甲基化水平、差异甲基化区域(DMRs)和CMT2靶向的转座元件(TEs)被多个GWAS定位。这与CMT2作为负责维持CHH甲基化的DNA甲基转移酶功能以及CHH和CHG甲基化之间的强相关性相吻合。另一个TEs上CHH甲基化的主要决定因子是NRPE1,它编码RNA聚合酶V的最大亚基,是RNA介导DNA甲基化(RdDM)通路的关键组分。NRPE1在调控TEs移动性方面的作用也通过GWAS揭示。
在一项GWAS中,miR823A通常与CHG DMRs相关。将CHH甲基化作为协变量后,另一项RdDM/CMT2靶向TEs的CHG甲基化的GWAS鉴定出CMT3和miR823A。CMT3编码负责维持CHG甲基化的DNA甲基转移酶,而miR823A编码的microRNA823A预计靶向CMT3。这些发现表明miR823A-CMT3模块介导的CHG甲基化调控可能,但还需要进一步验证。
包括AGO9和AGO1在内的ARGONAUTE基因被鉴定为自然群体中RdDM靶向TEs的CHH甲基化调控因子。这些基因在这些品系中的功能可能与Col-0中AGO9功能不同,因为ago9突变体在RdDM靶向的低甲基化区域(hypo-DMR)的甲基化没有变化。这表明在Col-0中没有参与RdDM通路的AGO蛋白可能在其他品系中参与DNA甲基化。
MULTICOPY SUPPRESSOR OF IRA1(MSI1)编码Polycomb抑制复合体2(PRC2)的一个亚基,该亚基催化组蛋白3赖氨酸27位点的三甲基化(H3K27me3)。在两项最近的GWAS中,MSI1与44个DMR区域的CHG甲基化以及RdDM和CMT2靶向的TEs相关。在Col-0背景下,H3K27me3与DNA甲基化无关,与CMT3对CHG甲基化所必需的H3K9me2不同。这些发现表明在其他品系中DNA甲基化和H3K27me3之间可能存在相关性,这有待进一步表征。
尽管没有被重复鉴定,JUMONJI26(JMJ26)—BONSAI METHYLATION 1(IBM1)的同源物基因突变被报告与使用CHH作为协变量的条件GWAS中RdDM靶向TEs的CHG甲基化相关联。敲除JMJ26导致RdDM靶向TEs的CHG甲基化增加。鉴定JMJ26的关键是在CHG甲基化的GWAS时将CHH甲基化作为协变量的条件分析。这一发现突出了GWAS在群体表观遗传学研究中的创新潜力,包括研究方法或群体构建。
除了转录调控因子外,大量的基因突变,无论是邻近还是远端,已被证明与靶位点的DNA甲基化变化相关联。尽管如此,关于这些关联背后的因果基因突变知之甚少,因为还没有验证这些关联背后的遗传变化。一些研究表明,诸如TE插入和结构变异(SVs)的基因突变可以影响附近位点的DNA甲基化。Ahmed等人(2011年)发现,没有匹配24-nt siRNA的数百个TEs通过邻近密集甲基化的TEs甲基化扩散获得DNA甲基化。另一项研究发现,在211个拟南芥品系中,近一半的新TE插入位点在带有TE插入的品系中高度甲基化并扩散到邻近区域。直接证据来自CEN180重复序列在常染色质靶位点的从头沉积,从而在ibm1突变体中诱导局部DNA甲基化的建立和扩散。在拟南芥1001个甲基化组中,大部分SVs(22%至50%)的甲基化存在差异,表明了SV对邻近序列DNA甲基化的影响。SV如何影响DNA甲基化的最好例子是拟南芥中的PHOSPHORIBOSYLANTHRANILATE ISOMERASE(PAI)基因家族(PAI1至PAI4),一些品系包含未甲基化的PAI基因,如Col,而其他品系包含甲基化的PAI基因,如WS。Col包含3个不连锁的PAI基因(PAI1、PAI2和PAI3),而WS包含4个PAI基因,具有PAI1至PAI4的倒置重复。这个倒置重复导致所有4个PAI基因甲基化。
易小结
最近的全基因组关联研究(GWASs)为理解转座元件(TEs)非CG甲基化的自然变异的遗传基础提供了重要信息。然而目前对CG甲基化的遗传基础以及其他基因组特征(如启动子区域和蛋白质编码基因)的DNA甲基化了解仍然知之甚少。高通量检测和验证表观遗传变化的功能,以及将这些变化在作物育种项目中的应用,是未来重要的研究工作。在群体水平上的包含多组学数据集的全基因组染色质图谱的可用性,无疑将加深对表观遗传变化的理解。结合大数据分析与表观遗传编辑技术,用于挖掘和应用表观遗传变化,将极大增强作物育种项目。
未解决的问题
- 表观遗传变化在植物抗性方面的作用有多大?
- 如何高通量检测和验证表观遗传变化功能?
- 如何有效地将表观遗传变化应用于作物育种项目?
Liu J, Zhong X. Population epigenetics: DNA methylation in the plant omics era. Plant Physiol. 2024 Feb 16. pii: 7609601. doi: 10.1093/plphys/kiae089. PubMed PMID: 38366882.





