儿童和成人实体瘤共有微小差异甲基化区域(mDMR)的全面分析
癌症是美国1~14岁儿童第二大常见死因,每年约有11000例新发病例和1200例死亡病例。与成人癌症相比,儿童肿瘤通常突变负荷较低。然而儿童肿瘤的表观基因组发生显著变化,尤其具有广泛的DNA甲基化变化。儿童肿瘤的罕见性对开发用于诊断、预后或治疗监测的癌症特异性生物标志物带来了重大挑战。
美国南加州大学Keck医学院转化基因组系Bodour Salhia团队研究了各种不同儿童肿瘤的共有DNA甲基化谱的潜力。研究在11种不同儿童癌症类型的31个复发性儿童肿瘤组织、13个邻近正常组织和20个血浆游离细胞(cell-free,cf)DNA样本上进行全基因组重亚硫酸盐测序(WGBS),定义了多种癌症类型样本间差异甲基化的最小焦点区域,称之为微小差异甲基化区域(minimally differentially methylated regions,mDMRs)。从公开数据库获取的 506 个儿童癌症样本和 5691 个成人癌症样本中也观察到这些甲基化变化,使用靶向杂交探针捕获法分析的44个儿童癌症样本中同样观察到这些甲基化变化。本研究最后表明了这些甲基化变化可在cfDNA中检测到,并可能作为早期检测或微小残留疾病的潜在cfDNA甲基化生物标志物。相关研究成果于2024年6月1日以“A comprehensive analysis of minimally differentially methylated regions common to pediatric and adult solid tumors”为题发表在《npj Precision Oncology》(IF 6.8)期刊上。

研究思路设计
测序分析:对儿童癌症进行WGBS测序分析(包括来自27名个体的31个肿瘤组织、13个正常组织和20个cfDNA样本,涵盖11种不同的儿童癌症亚型)
数据整合:通过不同肿瘤类型的数据整合,鉴定在多种癌症类型中样本数量最多的微小差异甲基化区域(mDMRs)。
下游验证:
(1)mDMRs 在TARGET公开数据库中的506个儿童癌症样本中(涵盖4种癌症类型)、
TCGA数据中的5691个成人癌症样本(涵盖14种癌症类型)、Capper等人的几种中枢神经系统肿瘤类型中均观察到。
(2)使用靶向杂交探针捕获检测法在一组独立的44个儿童癌症组织样本中进行进一步验证(来自6种肿瘤类型)。
结果图形
(1)分析儿童癌症的差异DNA甲基化模式
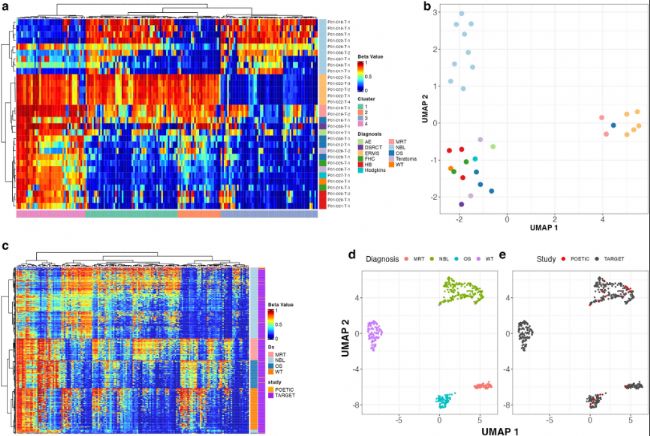
b) 基于183个最高变化DMRs样本进行均匀流形近似投影(UMAP)分析,如c所示。
c) 506个TARGET项目样本中166个区域和17个POETIC项目样本中183个区域的beta值热图(A中的17个区域由于450K芯片限制而被排除)。
d) 根据诊断和来源,对C中的区域进行TARGET和POETIC样本UMAP分析。
e) 分析TARGET和POETIC样本来源。
(2)不同肿瘤类型共有的DNA甲基化变化
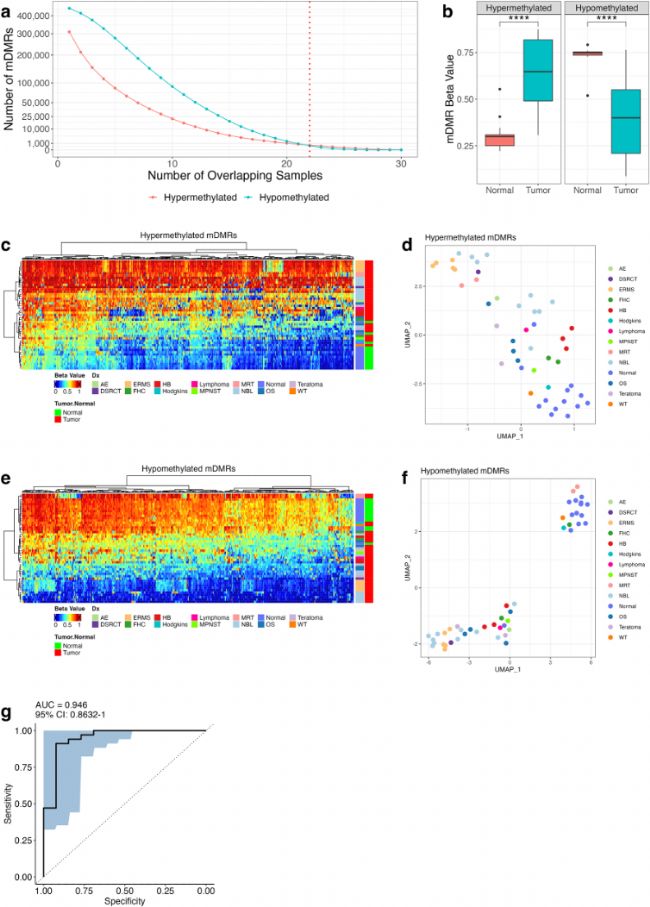
b) 低甲基化和高甲基化mDMRs的平均beta值箱线图。
c) 根据所有POETIC样本中所有低甲基化mDMR区域的beta值热图。
d) 基于所有POETIC样本中所有低甲基化mDMR区域的beta值UMAP分析。
e) 根据所有POETIC样本中所有高甲基化mDMR区域的beta值热图。
f) 基于所有POETIC样本中所有高甲基化mDMR区域的beta值UMAP。
g) 使用mDMRs构建的随机森林分类器模型的接收者操作特征(ROC)曲线。
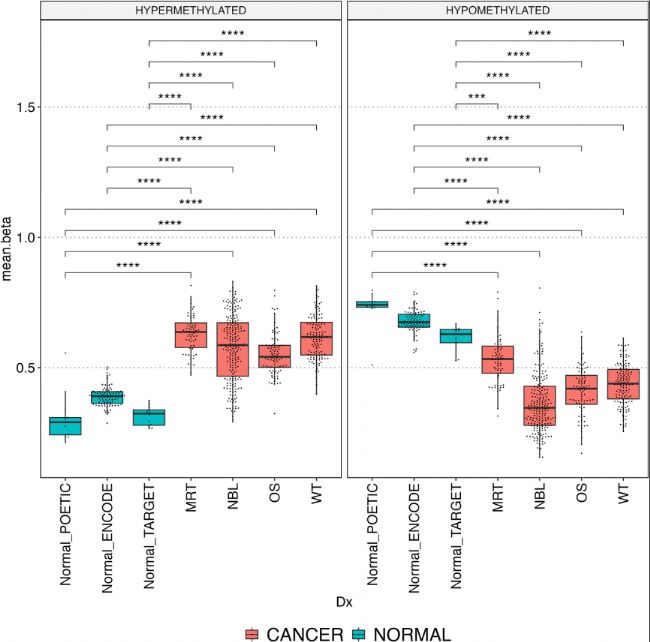
POETIC中的正常组织和ENCODE中的正常组织作为对照;所有比较均具有统计学意义(Wilcox p值≤0.0001),与POETIC样本中观察到的方向性一致。
(3)儿童癌症的mDMR也可用于检测成人癌症
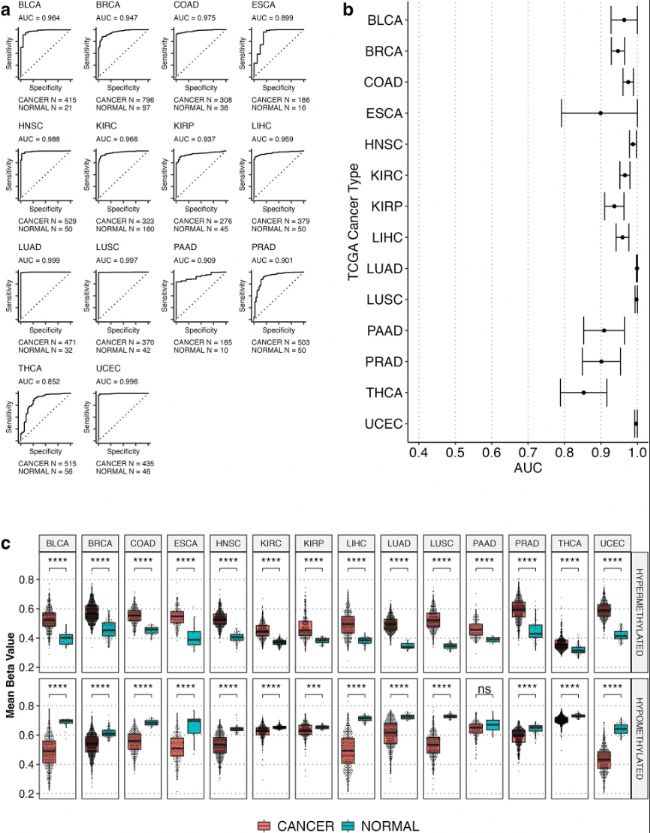
图4:TCGA数据库中多种成人癌症中检测到的mDMRs分析
a) WGBS来源的905个儿童癌症mDMRs中的422个(由于450K芯片限制,只使用422个子集)训练的随机森林模型,基于TCGA的450K DNA甲基化数据绘制的ROC曲线。
b) a中的AUC值图形,并以误差线的形式表示了95%置信区间(CI)。
c) 所有14种成人癌症类型中,高甲基化(上半部分)和低甲基化(下半部分)mDMRs的每样本肿瘤和正常组织的平均beta值箱线图。

图5:匹配的血浆和组织样本之间重叠的DMR。
a. cfDNA的总DMR。
b. cfDNA中检测到的DMRs与患者匹配的肿瘤组织样本中检测到的DMRs重叠的百分比。
c. 患者匹配的组织和cfDNA之间相交DMRs的差异甲基化的相关性图。
(5)泛儿童癌症(pan-pediatric)中共同存在且可检测的mDMRs,且这些mDMRs可以在cfDNA中被检测到
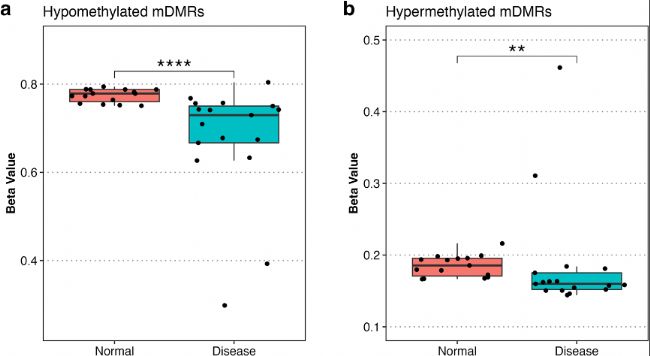
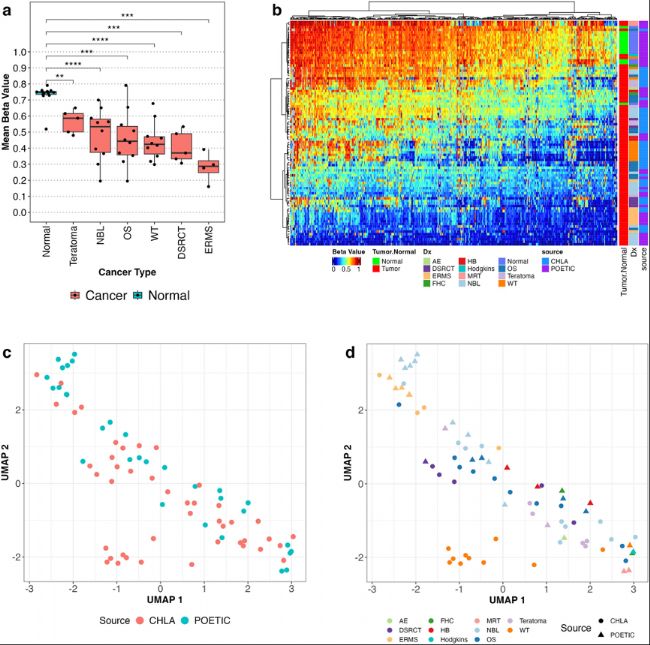
图 7:在独立的儿科癌症组织队列中对低甲基化 mDMR 进行靶向甲基化分析。
(6)在神经母细胞瘤(儿童常见实体瘤)样本中发现了广泛的特异性低甲基化区域,称为“DNA甲基化荒漠”
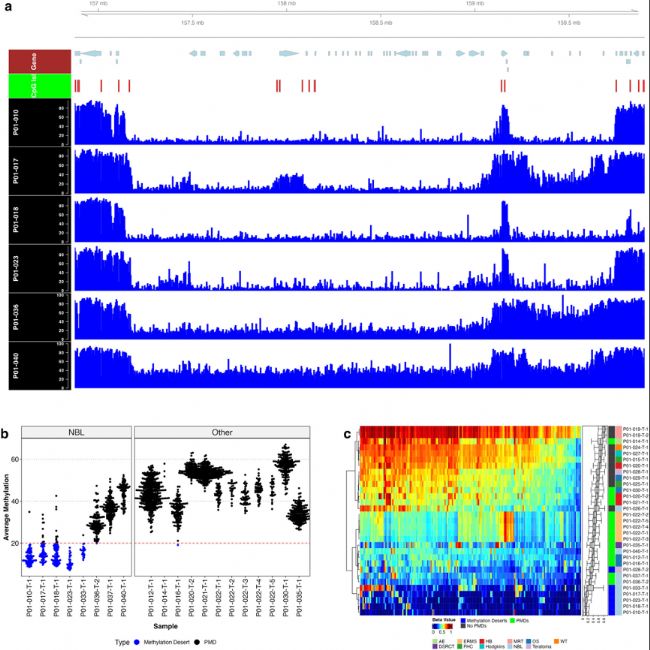 图8:通过全基因组亚硫酸盐测序(WGBS)技术揭示了神经母细胞瘤中两种显著的甲基化模式:DNA甲基化荒漠和经典的部分甲基化域(PMDs)
图8:通过全基因组亚硫酸盐测序(WGBS)技术揭示了神经母细胞瘤中两种显著的甲基化模式:DNA甲基化荒漠和经典的部分甲基化域(PMDs)
(7)WGBS在肿瘤组织gDNA中的拷贝数预估
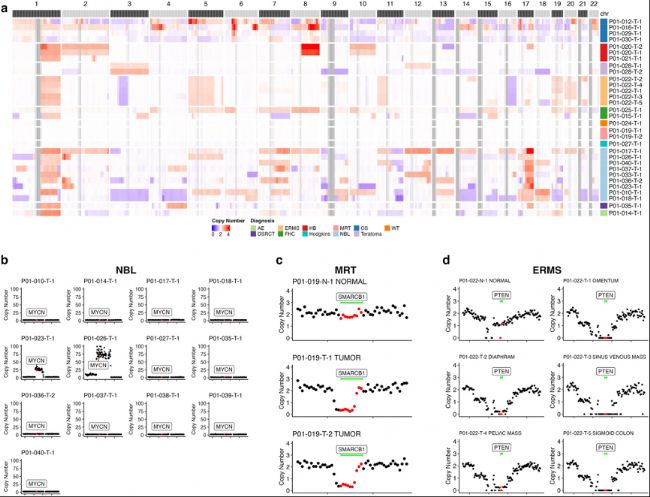
图9: WGBS在小儿癌症组织中的拷贝数评估。
(8)cfDNA中CNV检测的灵敏度低于cfDNA甲基化
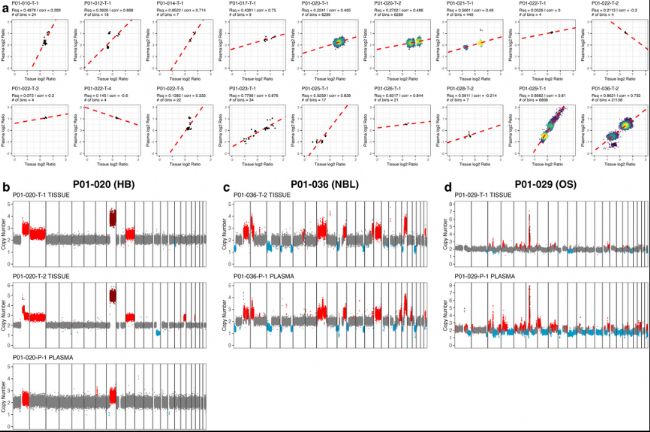
图10:cfDNA中CNVs的检测。
最终研究结果揭示了儿童肿瘤和成人肿瘤中的甲基化变化在cfDNA中可检测到,并可能作为潜在的cfDNA甲基化生物标志物。
儿童癌症DNA甲基化panel的鉴定可能为早期检测、治疗反应或微小残留疾病(MRD)的cfDNA甲基化液体活检提供基础。本研究揭示的甲基化模式既具有科学价值,也具有临床价值,旨在将这些分子见解转化为儿童和成人癌症的实际进展。
参考文献:
Buckley DN, Tew BY, Gooden C, Salhia B. A comprehensive analysis of minimally differentially methylated regions common to pediatric and adult solid tumors. NPJ Precis Oncol. 2024 Jun 1;8(1):125. pii: 10.1038/s41698-024-00590-1. doi: 10.1038/s41698-024-00590-1. PubMed PMID: 38824198.
美国南加州大学Keck医学院转化基因组系Bodour Salhia团队研究了各种不同儿童肿瘤的共有DNA甲基化谱的潜力。研究在11种不同儿童癌症类型的31个复发性儿童肿瘤组织、13个邻近正常组织和20个血浆游离细胞(cell-free,cf)DNA样本上进行全基因组重亚硫酸盐测序(WGBS),定义了多种癌症类型样本间差异甲基化的最小焦点区域,称之为微小差异甲基化区域(minimally differentially methylated regions,mDMRs)。从公开数据库获取的 506 个儿童癌症样本和 5691 个成人癌症样本中也观察到这些甲基化变化,使用靶向杂交探针捕获法分析的44个儿童癌症样本中同样观察到这些甲基化变化。本研究最后表明了这些甲基化变化可在cfDNA中检测到,并可能作为早期检测或微小残留疾病的潜在cfDNA甲基化生物标志物。相关研究成果于2024年6月1日以“A comprehensive analysis of minimally differentially methylated regions common to pediatric and adult solid tumors”为题发表在《npj Precision Oncology》(IF 6.8)期刊上。

研究思路设计
测序分析:对儿童癌症进行WGBS测序分析(包括来自27名个体的31个肿瘤组织、13个正常组织和20个cfDNA样本,涵盖11种不同的儿童癌症亚型)
数据整合:通过不同肿瘤类型的数据整合,鉴定在多种癌症类型中样本数量最多的微小差异甲基化区域(mDMRs)。
下游验证:
(1)mDMRs 在TARGET公开数据库中的506个儿童癌症样本中(涵盖4种癌症类型)、
TCGA数据中的5691个成人癌症样本(涵盖14种癌症类型)、Capper等人的几种中枢神经系统肿瘤类型中均观察到。
(2)使用靶向杂交探针捕获检测法在一组独立的44个儿童癌症组织样本中进行进一步验证(来自6种肿瘤类型)。
结果图形
(1)分析儿童癌症的差异DNA甲基化模式

图1:儿童癌症中差异DNA甲基化模式分析
a) 31个肿瘤样本中183个高度变化的差异化甲基化区域(DMRs)的beta值分层聚类。行注释条表示诊断结果;列注释条表示通过k均值聚类确定的DMRs聚类。b) 基于183个最高变化DMRs样本进行均匀流形近似投影(UMAP)分析,如c所示。
c) 506个TARGET项目样本中166个区域和17个POETIC项目样本中183个区域的beta值热图(A中的17个区域由于450K芯片限制而被排除)。
d) 根据诊断和来源,对C中的区域进行TARGET和POETIC样本UMAP分析。
e) 分析TARGET和POETIC样本来源。
(2)不同肿瘤类型共有的DNA甲基化变化

图2:WGBS分析揭示儿童癌症的共有DNA甲基化panel。
a) 不同数量样本(x轴)中重叠mDMRs的数量(y轴)。根据方向性,浅蓝色代表低甲基化区域,红色代表高甲基化区域。红色的虚线垂直线表示样本数量的截断点(22或更多样本,即70%肿瘤样本),由此产生402个低甲基化和503个高甲基化mDMRs。b) 低甲基化和高甲基化mDMRs的平均beta值箱线图。
c) 根据所有POETIC样本中所有低甲基化mDMR区域的beta值热图。
d) 基于所有POETIC样本中所有低甲基化mDMR区域的beta值UMAP分析。
e) 根据所有POETIC样本中所有高甲基化mDMR区域的beta值热图。
f) 基于所有POETIC样本中所有高甲基化mDMR区域的beta值UMAP。
g) 使用mDMRs构建的随机森林分类器模型的接收者操作特征(ROC)曲线。

图3:TARGET和ENCODE数据集中的mDMR。
方框图显示了TARGET数据中MRT、NBL OS和WT肿瘤样本中高甲基化(左)和低甲基化(右)mDMR的每个样本平均β值分布。每个点代表一个样本,中位数β和四分位数间距由方框图表示。POETIC中的正常组织和ENCODE中的正常组织作为对照;所有比较均具有统计学意义(Wilcox p值≤0.0001),与POETIC样本中观察到的方向性一致。
(3)儿童癌症的mDMR也可用于检测成人癌症

图4:TCGA数据库中多种成人癌症中检测到的mDMRs分析
b) a中的AUC值图形,并以误差线的形式表示了95%置信区间(CI)。
c) 所有14种成人癌症类型中,高甲基化(上半部分)和低甲基化(下半部分)mDMRs的每样本肿瘤和正常组织的平均beta值箱线图。

图5:匹配的血浆和组织样本之间重叠的DMR。
b. cfDNA中检测到的DMRs与患者匹配的肿瘤组织样本中检测到的DMRs重叠的百分比。
c. 患者匹配的组织和cfDNA之间相交DMRs的差异甲基化的相关性图。
(5)泛儿童癌症(pan-pediatric)中共同存在且可检测的mDMRs,且这些mDMRs可以在cfDNA中被检测到

图6:儿童癌症样本血浆mDMR的cfDNA甲基化分析。
a-b. 肿瘤样本中鉴定的402个低甲基化和503个高甲基化mDMR的cfDNA平均β值。代表了所有健康和癌症血浆样本的所有区域。
图 7:在独立的儿科癌症组织队列中对低甲基化 mDMR 进行靶向甲基化分析。
(6)在神经母细胞瘤(儿童常见实体瘤)样本中发现了广泛的特异性低甲基化区域,称为“DNA甲基化荒漠”

(7)WGBS在肿瘤组织gDNA中的拷贝数预估

图9: WGBS在小儿癌症组织中的拷贝数评估。
(8)cfDNA中CNV检测的灵敏度低于cfDNA甲基化

图10:cfDNA中CNVs的检测。
最终研究结果揭示了儿童肿瘤和成人肿瘤中的甲基化变化在cfDNA中可检测到,并可能作为潜在的cfDNA甲基化生物标志物。
儿童癌症DNA甲基化panel的鉴定可能为早期检测、治疗反应或微小残留疾病(MRD)的cfDNA甲基化液体活检提供基础。本研究揭示的甲基化模式既具有科学价值,也具有临床价值,旨在将这些分子见解转化为儿童和成人癌症的实际进展。
参考文献:
Buckley DN, Tew BY, Gooden C, Salhia B. A comprehensive analysis of minimally differentially methylated regions common to pediatric and adult solid tumors. NPJ Precis Oncol. 2024 Jun 1;8(1):125. pii: 10.1038/s41698-024-00590-1. doi: 10.1038/s41698-024-00590-1. PubMed PMID: 38824198.
标签:
DNA甲基化





