GlycanFinder:结合数据库搜索和从头测序的糖蛋白组学分析
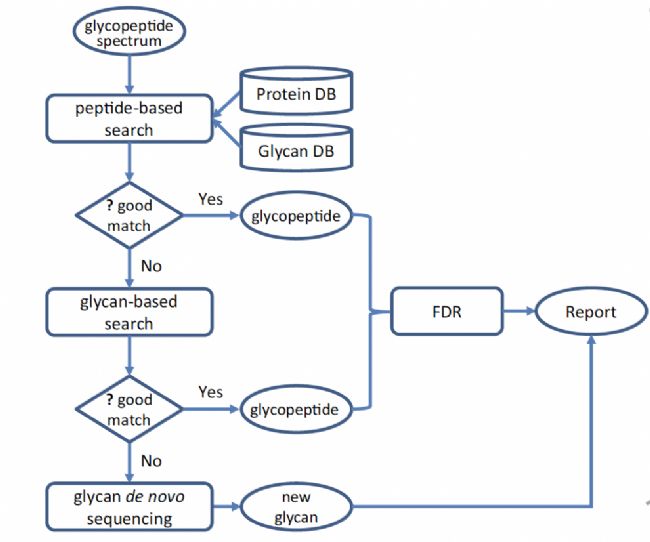
近日,来自于加拿大Bioinformatics Solutions Inc.的PEAKS团队、滑铁卢大学David R. Cheriton计算机科学学院的李明教授团队以及百蓁生物的研究者,在Nature Communication发表了结合数据库搜索和从头测序实现高灵敏度糖蛋白组学分析的PEAKS GlycanFinder软件工具。GlycanFinder集成了基于肽段和基于聚糖,二者兼顾的搜索策略,以解决来自糖肽极为复杂的碎片离子的解析问题。通过深度学习策略对聚糖复杂的分枝结构及其产生的碎片离子进行聚糖的从头测序。基于这样的策略,GlycanFinder还可以鉴定现有数据库中没有发现的糖肽。

随着液相与串联质谱(LC-MS /MS)的技术发展,糖蛋白组学中的研究中,已经能够对完整的糖肽进行位点特异性糖谱学分析,包括蛋白质、修饰位点、 聚糖结构,并可对其进行定量分析。但是对于糖肽鉴定仍然存在的挑战,糖肽产生的谱图非常的复杂,碎片离子类型众多,包括了来自于多肽的b/y,c/z,来自于聚糖的B/Y等。对于N-link和O-link的糖肽分析,同时考虑谱图中所呈现的肽段碎片信息或者聚糖碎片信息,基于蛋白和聚糖的数据库挑选出所有可能的候选。当来自于多肽的信号或者聚糖的信号较差时,造成难以挑选出候选糖肽的不利局面,则可被改善。另外,作者通过整合了动态规划,图形神经网络,Transformer神经网络构建了学习糖结构和基于质谱数据重建聚糖分枝结构的深度学习模型。并且对于糖肽的鉴定结果,从多肽水平和聚糖水平分别通过Target-Decoy的策略进行FDR的评估。
1. 完整糖肽分析的工作流程
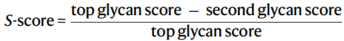 2. 基于深度学习的N-link糖肽从头测序
2. 基于深度学习的N-link糖肽从头测序
Glycan从头测序的评估
3. 多肽和聚糖的FDR评估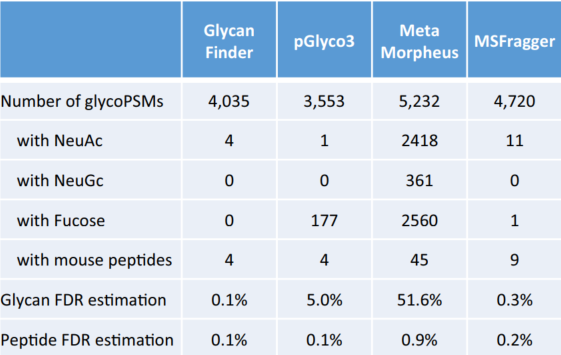

5. O-连接糖肽分析方法的评价
O-连接糖是通过丝氨酸(S)或苏氨酸(T)残基的羟基连接到蛋白上的。因此,在一个肽序列中通常有不止一个的O-糖基化位点。GlycanFinder允许每个肽最多两个O-连接的聚糖,并使用内部碎片离子来确定最佳糖基化位点,计算其糖基化位点分配的特定位点定位分数(A-Score)。
GlycanFinder的总体得分为0.730,在超过了Kawahara等人之前的研究中,其他9个软件报告的最好结果。总体而言,基于HGI研究的benchmark结果表明,GlycanFinder代表了N-糖蛋白组学和O-糖蛋白组学的高性能信息解决方案。
参考文献
Sun, W., Zhang, Q., Zhang, X. et al. Glycopeptide database search and de novo sequencing with PEAKS GlycanFinder enable highly sensitive glycoproteomics. Nat Commun 14, 4046 (2023). https://doi.org/10.1038/s41467-023-39699-5
悄悄剧透一下,GlycanFinder 2.0 版本即将发布,敬请期待! (点击图片即可查看活动详情)
如果您想深入了解更多关于PEAKS 软件更多内容,欢迎扫描下方二维码关注我们!

随着液相与串联质谱(LC-MS /MS)的技术发展,糖蛋白组学中的研究中,已经能够对完整的糖肽进行位点特异性糖谱学分析,包括蛋白质、修饰位点、 聚糖结构,并可对其进行定量分析。但是对于糖肽鉴定仍然存在的挑战,糖肽产生的谱图非常的复杂,碎片离子类型众多,包括了来自于多肽的b/y,c/z,来自于聚糖的B/Y等。对于N-link和O-link的糖肽分析,同时考虑谱图中所呈现的肽段碎片信息或者聚糖碎片信息,基于蛋白和聚糖的数据库挑选出所有可能的候选。当来自于多肽的信号或者聚糖的信号较差时,造成难以挑选出候选糖肽的不利局面,则可被改善。另外,作者通过整合了动态规划,图形神经网络,Transformer神经网络构建了学习糖结构和基于质谱数据重建聚糖分枝结构的深度学习模型。并且对于糖肽的鉴定结果,从多肽水平和聚糖水平分别通过Target-Decoy的策略进行FDR的评估。
1. 完整糖肽分析的工作流程
为了提高灵敏度,GlycanFinder同时应用基于肽和糖的搜索。如果在基于多肽的搜索后谱图仍未被鉴定,则会进行基于聚糖的搜索。基于肽和聚糖搜索的组合利用了肽和聚糖碎片离子,因此减少了因为碎片信号差而错过一些候选的概率。确定了候选糖肽,在第二轮打分时,会综合考虑肽主链产生的离子、糖肽 Y 离子和 B 离子评估糖肽谱匹配 (glycoPSM) 并估计其 FDR。

糖肽分析中的一个常见问题是,在一个肽序列中可能存在多个糖基化位点或具有相同组成的多个异构体的聚糖,这增加了糖肽匹配的不确定性。通过A-score计算位点特异性,由排名第一和第二候选位点的分数差获得。类似地,当具有相同组成的多个异构体聚糖匹配一个谱图时,它们的结构分数是用它们各自的糖肽Y离子来计算的。通过排名第一和排名第二的糖结构分数差来计算S-score,然后选择得分最高的多糖。glycoPSM的A-score和S-score反映了其糖基化位点和糖链结构分配的置信度,因为第一和第二候选得分之间的较大差异意味着最好得分(方法)的分配得到了更有力的支持证据。
glycoPSM的S得分定义为:
glycoPSM的S得分定义为:
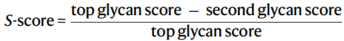
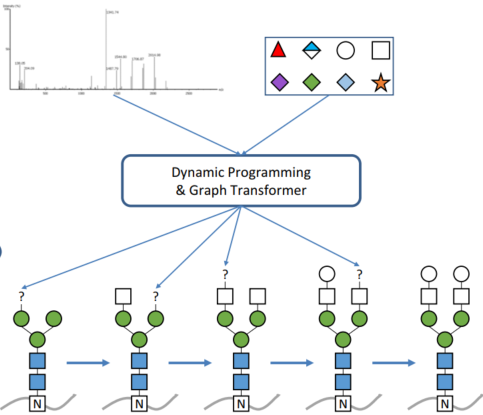
与先前报道的一些聚糖从头测序的方法不同,GlycanFinder遵循数据驱动的方法,并应用机器学习模型从训练数据中学习聚糖结构。
给定一张谱图以及聚糖的质量数(通过母离子质量数-多肽质量数),作者通过动态规划的方法计算糖的组成,树状的糖结构从“根”到“叶”进行构建。深度学习模型用于预测下一个单糖。将五种单糖(Hex、HexNAc、Fuc、NeuAc、NeuGc)或其可能产生的每一种组合添加到部分树中创建候选树池,然后用两个神经网络(其中一个Graphormer神经网络用来捕获候选树的结构,另一个用来捕获候选树和谱图之间匹配糖肽的Y,B离子)对每个候选树进行评分,选择得分最高的树进行下一次迭代。直到由动态规划计算出的聚糖组成的所有单糖均已被使用。该模型的核心思想和多肽从头测序一样,希望借由 Transformer 图神经网络捕获的候选树的结构,在碎片离子之外提供额外的证据来预测下一个单糖。
给定一张谱图以及聚糖的质量数(通过母离子质量数-多肽质量数),作者通过动态规划的方法计算糖的组成,树状的糖结构从“根”到“叶”进行构建。深度学习模型用于预测下一个单糖。将五种单糖(Hex、HexNAc、Fuc、NeuAc、NeuGc)或其可能产生的每一种组合添加到部分树中创建候选树池,然后用两个神经网络(其中一个Graphormer神经网络用来捕获候选树的结构,另一个用来捕获候选树和谱图之间匹配糖肽的Y,B离子)对每个候选树进行评分,选择得分最高的树进行下一次迭代。直到由动态规划计算出的聚糖组成的所有单糖均已被使用。该模型的核心思想和多肽从头测序一样,希望借由 Transformer 图神经网络捕获的候选树的结构,在碎片离子之外提供额外的证据来预测下一个单糖。

Glycan从头测序的评估
基于五种小鼠组织(脑、心脏、肾、肝、肺)的数据集,作者评估了聚糖从头测序的深度学习模型。首先进行N-link糖库搜索,并以 1% FDR 识别出 139,208 个 glycoPSM,随后用于训练和测试(以五次交叉验证方式进行,其中四种组织的 glycoPSM 用于训练,其余组织的 glycoPSM 用于测试),例如肺的数据用与测试时,其他四种(脑、肾、心、肝脏)的数据用于训练。从训练集中排除了测试集的所有聚糖,以确保训练集和测试集没有任何共同的聚糖。
对于每个 glycoPSM,de novo 聚糖与目标聚糖(从数据库搜索中识别)基于三个级别:组成、碎片离子和结构进行比较。
第一级的评估在于de novo 和目标的糖是否具有相同的组成,第二,从头测序的糖和目标的糖匹配的糖碎片离子的数量,第三,计算de novo和目标糖结构是否完全匹配。
GlycanFinder 在结构,碎片离子和组成三个层次上的平均准确度分别为 32%、83% 和 89%,而 StrucGP 的准确度分别为 23%、84% 和 85%。虽然这两种软件显示的碎片离子和成分的准确性相当,但 GlycanFinder 的平均结构准确性大大高于 StrucGP,在所有五种组织中都是如此。结果证明GlycanFinder 深度学习模型在聚糖从头测序的树结构学习和预测方面具有优势。
对于每个 glycoPSM,de novo 聚糖与目标聚糖(从数据库搜索中识别)基于三个级别:组成、碎片离子和结构进行比较。
第一级的评估在于de novo 和目标的糖是否具有相同的组成,第二,从头测序的糖和目标的糖匹配的糖碎片离子的数量,第三,计算de novo和目标糖结构是否完全匹配。
GlycanFinder 在结构,碎片离子和组成三个层次上的平均准确度分别为 32%、83% 和 89%,而 StrucGP 的准确度分别为 23%、84% 和 85%。虽然这两种软件显示的碎片离子和成分的准确性相当,但 GlycanFinder 的平均结构准确性大大高于 StrucGP,在所有五种组织中都是如此。结果证明GlycanFinder 深度学习模型在聚糖从头测序的树结构学习和预测方面具有优势。
3. 多肽和聚糖的FDR评估
对于多肽的FDR评估,一般采用标准的target-decoy方法,通过对目标库蛋白序列进行随机打散的方式构建诱饵库蛋白。而对于聚糖的 FDR,由于其非线性结构,则应用碎片离子的质量随机偏移来创建诱饵谱图。仅当 glycoPSM 的肽和聚糖 FDR 均小于或等于 1% 时,它才能通过 1% FDR 阈值。
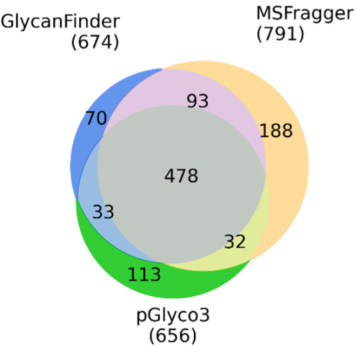
通过对裂殖酵母糖蛋白组样本数据集进行 N糖肽分析,对GlycanFinder,pGlyco3(版本 20210615)、MetaMorpheus(版本 0.0.320)和 MSFragger(版本 19.0)进行FDR评估后的对比。所有软件均使用相同的数据库和参数。结果如图所示,GlycanFinder 鉴定出 4035 个 glycoPSMs,比pGlyco3 (3553)多13.6%,比 MSFragger (4720) 低 17.0%。MetaMorpheus 鉴定最多glycoPSM(5232),但它FDR高达 51.6%。当GlycanFinder的Glycan 和Peptide FDR阈值放宽,调整为 0.3% 和 0.2%,以便GlycanFinder与MSFragger的 FDR进行比较,此时GlycanFinder 鉴定出 4518 个 glycoPSM,比MSFragger少4.5%。但值得注意的是MSFragger报告的是聚糖成分,而 GlycanFinder 和 pGlyco3 报告的是聚糖结构,可提供聚糖更全面的信息。
通过对裂殖酵母糖蛋白组样本数据集进行 N糖肽分析,对GlycanFinder,pGlyco3(版本 20210615)、MetaMorpheus(版本 0.0.320)和 MSFragger(版本 19.0)进行FDR评估后的对比。所有软件均使用相同的数据库和参数。结果如图所示,GlycanFinder 鉴定出 4035 个 glycoPSMs,比pGlyco3 (3553)多13.6%,比 MSFragger (4720) 低 17.0%。MetaMorpheus 鉴定最多glycoPSM(5232),但它FDR高达 51.6%。当GlycanFinder的Glycan 和Peptide FDR阈值放宽,调整为 0.3% 和 0.2%,以便GlycanFinder与MSFragger的 FDR进行比较,此时GlycanFinder 鉴定出 4518 个 glycoPSM,比MSFragger少4.5%。但值得注意的是MSFragger报告的是聚糖成分,而 GlycanFinder 和 pGlyco3 报告的是聚糖结构,可提供聚糖更全面的信息。

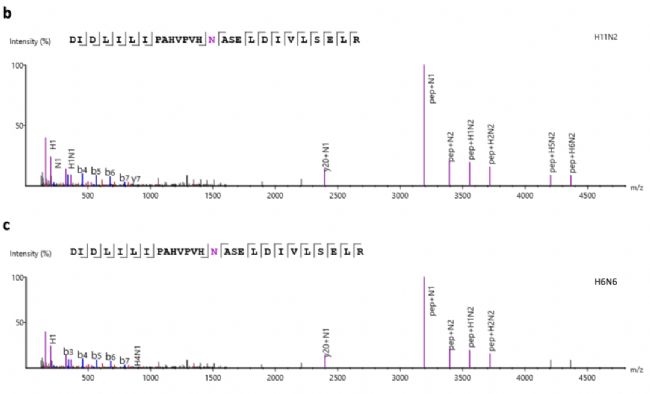
GlycanFinder还鉴定到70种别的软件没有鉴定到的糖肽。如图所示,在蛋白质PO13781|YEO3_SCHPO上的糖基化位点N234处鉴定到的glycoPSMs。GlycanFinder 在该位点鉴定了三个裂殖酵母中常见的聚糖,包括具有高甘露糖结构的 (HexNAc)2(Hex)11 和 (HexNAc)2(Hex)13 ( HexNAc)2(Hex)n。并且在样品2的编号56,805谱图中,GlycanFinder在同样谱图上鉴定到的高甘露糖聚糖 H11N2比pGlyco3鉴定的H6N6有更好的糖肽B/Y离子。而MSFragger 则没有在该糖基化位点的报告任何鉴定结果。


4. 基于HUPO人类糖蛋白组学计划的研究综合评估
Kawahara等人最近描述了HUPO 人类糖蛋白组学计划 (HGI)研究,评估来自9个开发团队和13个用户团队的11个糖蛋白组学软件的性能,以进行完整的糖肽分析。
他们的研究提供了来自人类血清的标准糖蛋白组学数据集和全面的评价标准。在这里,我们还在相同的基准上对GlycanFinder进行了评估,并与Kawahara报告的结果进行了比较,研究中的结果包括IQ-GPA v2.5,Prospector v5.20.23,GlyXtoolMS v0.1.4,Byonic v2.16.16,Sugar Qb,Glycopeptide Search v2.0alpha、GlyCopeptideGraphMS v1.0、GlycoPAT v2.0和GPQuest v2.0。我们还将我们的结果与Kawahara等人报告的最佳用户团队的结果进行了比较。
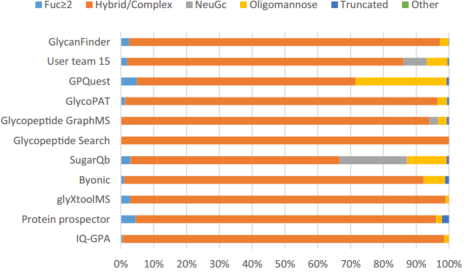
结果显示,总体而言,GlycanFinder的性能略好于报告的最佳结果 (0.789 vs 0.777),并超过了其他九个软件。此外,GlycanFinder在N1-N3、N5、N6(0.833-0.952)的五个标准中始终得分很高,鉴定N-连接糖肽和糖蛋白方面具有很高的准确性,同时适当地控制了FDR。然而,在N4测试中,GlycanFinder在识别的N-连接糖肽的数量方面表现不佳。我们进一步检查了GlycanFinder和其他工具的结果之间的一致性。左图显示GlycanFinder的83%的N-连接的糖链成分和78%的N-连接的糖蛋白也被至少三个其他工具报告。右图显示了GlycanFinder报告的N-连接聚糖与其他高评分工具(如User Team 15、Prospector或Byonic)报告的N-连接聚糖的一致分类。
Kawahara等人最近描述了HUPO 人类糖蛋白组学计划 (HGI)研究,评估来自9个开发团队和13个用户团队的11个糖蛋白组学软件的性能,以进行完整的糖肽分析。
他们的研究提供了来自人类血清的标准糖蛋白组学数据集和全面的评价标准。在这里,我们还在相同的基准上对GlycanFinder进行了评估,并与Kawahara报告的结果进行了比较,研究中的结果包括IQ-GPA v2.5,Prospector v5.20.23,GlyXtoolMS v0.1.4,Byonic v2.16.16,Sugar Qb,Glycopeptide Search v2.0alpha、GlyCopeptideGraphMS v1.0、GlycoPAT v2.0和GPQuest v2.0。我们还将我们的结果与Kawahara等人报告的最佳用户团队的结果进行了比较。
结果显示,总体而言,GlycanFinder的性能略好于报告的最佳结果 (0.789 vs 0.777),并超过了其他九个软件。此外,GlycanFinder在N1-N3、N5、N6(0.833-0.952)的五个标准中始终得分很高,鉴定N-连接糖肽和糖蛋白方面具有很高的准确性,同时适当地控制了FDR。然而,在N4测试中,GlycanFinder在识别的N-连接糖肽的数量方面表现不佳。我们进一步检查了GlycanFinder和其他工具的结果之间的一致性。左图显示GlycanFinder的83%的N-连接的糖链成分和78%的N-连接的糖蛋白也被至少三个其他工具报告。右图显示了GlycanFinder报告的N-连接聚糖与其他高评分工具(如User Team 15、Prospector或Byonic)报告的N-连接聚糖的一致分类。


5. O-连接糖肽分析方法的评价
O-连接糖是通过丝氨酸(S)或苏氨酸(T)残基的羟基连接到蛋白上的。因此,在一个肽序列中通常有不止一个的O-糖基化位点。GlycanFinder允许每个肽最多两个O-连接的聚糖,并使用内部碎片离子来确定最佳糖基化位点,计算其糖基化位点分配的特定位点定位分数(A-Score)。
GlycanFinder的总体得分为0.730,在超过了Kawahara等人之前的研究中,其他9个软件报告的最好结果。总体而言,基于HGI研究的benchmark结果表明,GlycanFinder代表了N-糖蛋白组学和O-糖蛋白组学的高性能信息解决方案。
参考文献
Sun, W., Zhang, Q., Zhang, X. et al. Glycopeptide database search and de novo sequencing with PEAKS GlycanFinder enable highly sensitive glycoproteomics. Nat Commun 14, 4046 (2023). https://doi.org/10.1038/s41467-023-39699-5
悄悄剧透一下,GlycanFinder 2.0 版本即将发布,敬请期待! (点击图片即可查看活动详情)
如果您想深入了解更多关于PEAKS 软件更多内容,欢迎扫描下方二维码关注我们!







