序列数据库搜索评分基本原理详解
LDF评分
PEAKS DB中的评分计算如下图所示 PEAKS DB在内部使用LDF评分(线性判别函数)来评判肽谱图的匹配质量。LDF评分不仅使用碎片离子与谱图中碎片峰之间的匹配,还考虑许多其他因素,例如de novo测序的多肽和数据库搜索得到的多肽序列之间的相似性。
PEAKS DB在内部使用LDF评分(线性判别函数)来评判肽谱图的匹配质量。LDF评分不仅使用碎片离子与谱图中碎片峰之间的匹配,还考虑许多其他因素,例如de novo测序的多肽和数据库搜索得到的多肽序列之间的相似性。
LDF评分可以实现以下两个目标:
P-Value
LDF分数将转换为P值,以便更好地进行人工诠释。
P值:对于一个给定的评分x, 其相应的P值是“一个错误匹配得到的分值>x”的概率。
P值越小,肽-谱图匹配是随机匹配的概率就越小。下图更好地解释了P值的含义。 请注意,尽管许多软件包中都使用“P-value”,它们的含义可能各不相同。P值的另一个流行的定义是“肽段与当前谱图匹配得分>x是随机匹配的概率”。然而,在数据库搜索中,错误鉴定是数据库中许多随机肽的结果,而不仅仅是一个随机肽。因此,PEAKS DB中的P值定义对于控制结果的质量更加有用。
请注意,尽管许多软件包中都使用“P-value”,它们的含义可能各不相同。P值的另一个流行的定义是“肽段与当前谱图匹配得分>x是随机匹配的概率”。然而,在数据库搜索中,错误鉴定是数据库中许多随机肽的结果,而不仅仅是一个随机肽。因此,PEAKS DB中的P值定义对于控制结果的质量更加有用。
-10logP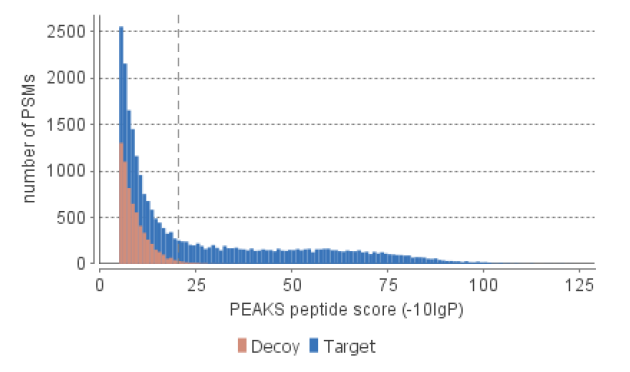
 (点击图片即可查看活动详情)
(点击图片即可查看活动详情)
如果您想深入了解更多关于PEAKS 软件更多内容,欢迎扫描下方二维码关注我们!
PEAKS DB中的评分计算如下图所示
LDF评分可以实现以下两个目标:
- 对于MS/MS数据集中的每个谱图,从数据库中找到最有可能正确的肽;
- 对于整个数据集,尽可能分出正确匹配与错误匹配。
P-Value
LDF分数将转换为P值,以便更好地进行人工诠释。
P值:对于一个给定的评分x, 其相应的P值是“一个错误匹配得到的分值>x”的概率。
P值越小,肽-谱图匹配是随机匹配的概率就越小。下图更好地解释了P值的含义。

-10logP
将P值转换为 -10*log10(P值),使其更加“人性化”。在PEAKS中,该值用-10lgP表示,因为lg是log10的ISO保留表示法。通过此转换,更显著的匹配将对应更高的-10lgP值。此外,P值为1% 时,即-10lgP 为 20。
下图是PEAKS数据库搜索结果的屏幕截图。x轴是 -10lgP 分数,y 轴是在该分数下的肽谱匹配数量。通常,大于20的分数具有相对较高的置信度(如图中所示有许多目标,但很少有诱饵匹配超过该阈值)。对于大型数据集,建议使用FDR(错误发现率)来选择正确的 -10lgP分数阈值(这在PEAKS中很容易)。但是,当数据集很小时(#谱图“<100或蛋白质数据库仅包含少量蛋白质),直接选择-10lgP=20是更合适的筛选方法。
下图是PEAKS数据库搜索结果的屏幕截图。x轴是 -10lgP 分数,y 轴是在该分数下的肽谱匹配数量。通常,大于20的分数具有相对较高的置信度(如图中所示有许多目标,但很少有诱饵匹配超过该阈值)。对于大型数据集,建议使用FDR(错误发现率)来选择正确的 -10lgP分数阈值(这在PEAKS中很容易)。但是,当数据集很小时(#谱图“<100或蛋白质数据库仅包含少量蛋白质),直接选择-10lgP=20是更合适的筛选方法。

参考文献
- Zhang J, Xin L, Shan B, Chen W, Xie M, Yuen D, Zhang W, Zhang Z, Lajoie G.A., Ma B, PEAKS DB: De Novo Sequencing Assisted Database Search for Sensitive and Accurate Peptide Identification. Mol. Cell. Proteomics. 11, M111.010587 (2012).
- Xin, L., Qiao, R., Chen, X. et al. A streamlined platform for analyzing tera-scale DDA and DIA mass spectrometry data enables highly sensitive immunopeptidomics. Nat Commun 13, 3108 (2022). doi:10.1038/s41467-022-30867-7
 (点击图片即可查看活动详情)
(点击图片即可查看活动详情)如果您想深入了解更多关于PEAKS 软件更多内容,欢迎扫描下方二维码关注我们!

标签:
定性分析;搜库





