PEAKS中的蛋白质翻译后修饰分析强大功能详解
蛋白质翻译后修饰(PTM),如磷酸化、泛素化、乙酰化和甲基化,在信号传导和调控过程、蛋白质活性和降解、基因表达调控等多种生物过程中起着关键作用。PTM的鉴定和表征对于全面了解细胞生物学和人类疾病至关重要,并且具有广泛的应用。由于蛋白质数据库中PTM信息通常不存在或不完整,因此通过质谱法鉴定PTM给传统的数据库搜索方法带来了许多挑战。
PEAKS中包含的高级算法可最大限度地提高PTM鉴定和PTM分析。PEAKS PTM的翻译后修饰鉴定是通过整合PEAKS数据库搜索和De Novo测序结果来实现的。PTM Profile工具通过提供定性和定量信息以及对结果的可视化和直接导出,进一步协助您的PTM研究。
PEAKS中的PTM分析
PEAKS中的PTM分析
- 通过PEAKS数据库搜索来鉴定PTM
- 通过PEAKS PTM发现非指定的或隐藏的修饰来达到最大程度的鉴定PTM
- 通过PEAKS PTM对PTM进行定量分析
通过LC-MS/MS进行翻译后修饰(PTM)分析

通过LC-MS/MS进行翻译后修饰(PTM)分析
PTM 鉴定
PTM鉴定工作可以通过PEAKS数据库搜索和PEAKS PTM搜索来完成。
01通过数据库搜索
在数据搜索的方法中,仅仅对于有限个数的一些常见翻译后修饰可以被定义为可变修饰。
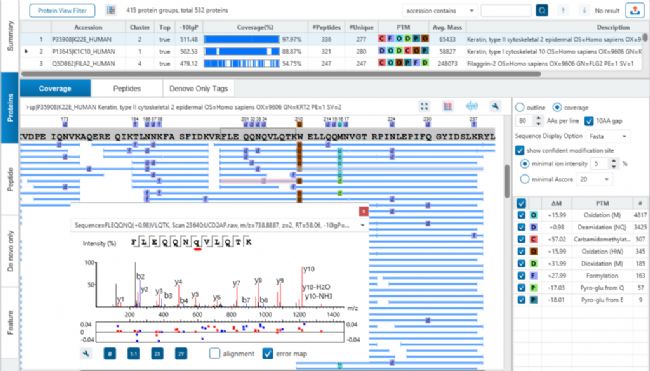
蛋白的结果视图中,鉴定到的支持性多肽证据以蓝色的条,映射到选中的蛋白的序列上。通过点击多肽,该多肽的二级质谱图将会显示出来,您可以很容易地检查质谱数据的注释。
在二级谱图的窗口中显示的多肽序列里,带有修饰的氨基酸残基以小写字母表示,例如在下图中,序列DTLmISR的甲硫氨酸。
蛋白的结果视图中,鉴定到的支持性多肽证据以蓝色的条,映射到选中的蛋白的序列上。通过点击多肽,该多肽的二级质谱图将会显示出来,您可以很容易地检查质谱数据的注释。
在二级谱图的窗口中显示的多肽序列里,带有修饰的氨基酸残基以小写字母表示,例如在下图中,序列DTLmISR的甲硫氨酸。
02通过PEAKS PTM 搜索
PEAKS PTM是整合了强大的de novo算法和数据库搜索算法的,特别为发现隐藏修饰而设计的功能模块。
这种多轮搜索方法在数据分析工作流图所描述:
01.对每一张谱图进行de novo测序
02.PEAKS数据库(DB)算法用于蛋白质鉴定。在这一轮中可以指定一些高频的常见PTM,以更好地提高灵敏度
03. PEAKS的PTM算法用于鉴定更多的翻译后修饰。在这一轮,仅仅通过de novo高置信度打分,并且同时没有在数据库中匹配的谱图被映射到已鉴定的蛋白上。用户可以定义任意多个的PTM,或者他们可以简单的运行一轮PTM搜索,可以实现650种Unimod数据库中翻译后修饰的同时检索。
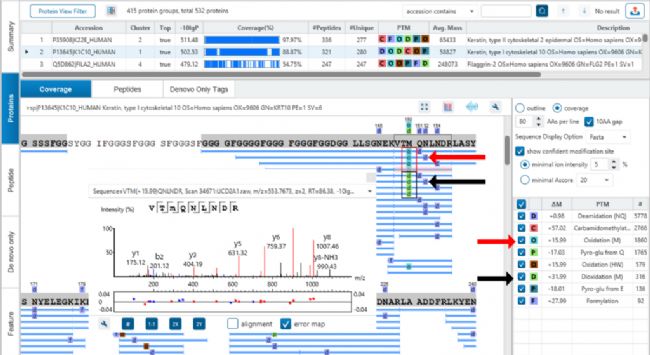
PEAKS PTM模块鉴定到另外的修饰,例如下图,除了在数据搜索得到的,在DTLMISR序列中甲硫氨酸的oxidation(图中黑色)以外,还有一种修饰dethiomethyl(图中红色指向)。
这种多轮搜索方法在数据分析工作流图所描述:
01.对每一张谱图进行de novo测序
02.PEAKS数据库(DB)算法用于蛋白质鉴定。在这一轮中可以指定一些高频的常见PTM,以更好地提高灵敏度
03. PEAKS的PTM算法用于鉴定更多的翻译后修饰。在这一轮,仅仅通过de novo高置信度打分,并且同时没有在数据库中匹配的谱图被映射到已鉴定的蛋白上。用户可以定义任意多个的PTM,或者他们可以简单的运行一轮PTM搜索,可以实现650种Unimod数据库中翻译后修饰的同时检索。
PEAKS PTM模块鉴定到另外的修饰,例如下图,除了在数据搜索得到的,在DTLMISR序列中甲硫氨酸的oxidation(图中黑色)以外,还有一种修饰dethiomethyl(图中红色指向)。

PTM 位点确证
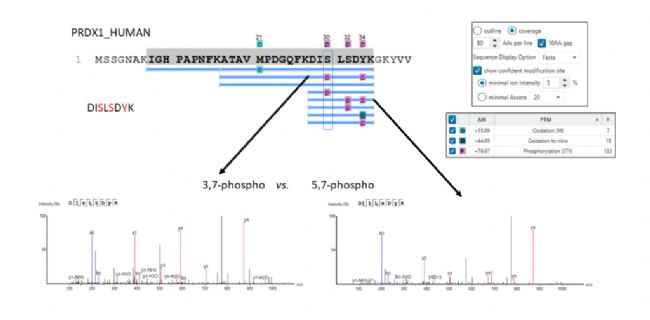
修饰的确切位置可由谱图中存在决定位点的碎片离子来确定。在下图所示的例子中,MS2谱图中b-11和b-12离子的存在决定了天冬酰胺在第12位发生脱氨基。
PEAKS为用户提供两个选择以确定可信的修饰位点:
01.最小离子强度,这要求MS/MS谱图中决定位置的碎片离子的相对强度必须高于用户输入的数值。
02.Ascore,将歧义分数计算为 -10 × log10 P。p 值表示肽偶然匹配的可能性。因此,Ascore越高置信度越高。
这两种方法都提供了PTM修饰位点定位的置信度。如果达到了用户选择方法的阈值,则PTM将显示在蛋白质覆盖率视图中残基上方的彩色框中。
PEAKS为用户提供两个选择以确定可信的修饰位点:
01.最小离子强度,这要求MS/MS谱图中决定位置的碎片离子的相对强度必须高于用户输入的数值。
02.Ascore,将歧义分数计算为 -10 × log10 P。p 值表示肽偶然匹配的可能性。因此,Ascore越高置信度越高。
这两种方法都提供了PTM修饰位点定位的置信度。如果达到了用户选择方法的阈值,则PTM将显示在蛋白质覆盖率视图中残基上方的彩色框中。

PTM 定量分析
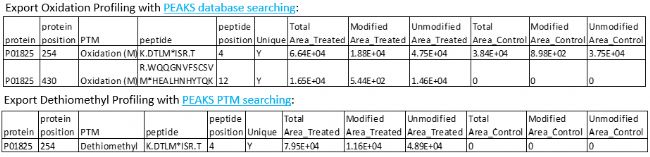
PEAKS PTM Profile提供了对定量信息直接地可视化和总结(例如,在鉴定到的位点,修饰和未修饰形式的丰度)。
PEAKS PTM Profile提供了对定量信息直接地可视化和总结(例如,在鉴定到的位点,修饰和未修饰形式的丰度)。
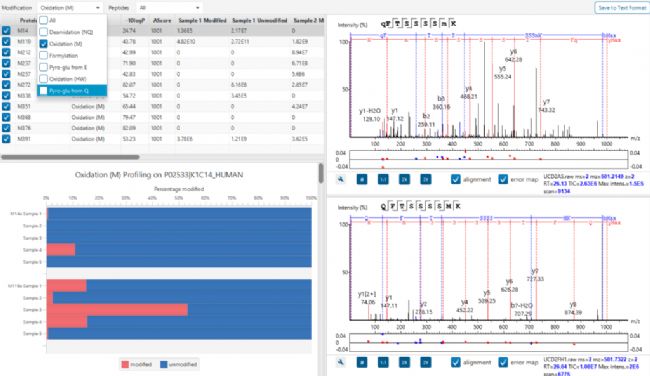
PTM profile的结果可以导出图像和包含有详细定量信息的CSV格式结果。
Reference
Han, X. et al. PEAKS PTM: Mass Spectrometry Based Identification of Peptides with
Unspecified Modifications. Journal of Proteomics Research. 10(7), 29302936. 24/05
/2011.
Unspecified Modifications. Journal of Proteomics Research. 10(7), 29302936. 24/05
/2011.
如果您想深入了解更多关于PEAKS的PTM分析,欢迎扫描下方二维码关注我们!






