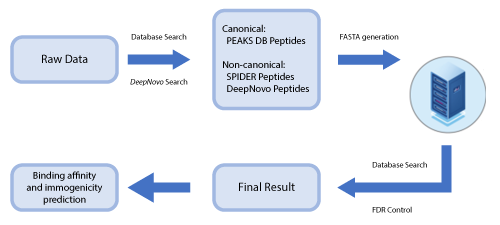
DeepNovo Peptidome一个新的肽组学专属工作流程
基于质谱的多肽组学已成为开发多肽疫苗和免疫疗法的热门方法。这一研究领域也导致了许多non-canonical多肽的发现,并改变了我们对基因组区域阅读和翻译的理解。在肽组学中,将正确的氨基酸序列分配到MS/MS谱图是至关重要的,因为每个残基的位置和鉴定结果直接影响多肽-蛋白质的相互作用,并确定它是否是进一步完成下游分析的优质候选。随着多肽组学分析和免疫肽组分析的普及,迫切需要有一个软件工具以准确和直观的方式有效地处理这类数据。
在多肽组学数据的质谱分析中,PEAKS软件已成为首选的数据分析解决方案,作为PEAKS Studio 11和PEAKS Online 11的一部分,我们提供了一个新的DeepNovo Peptidome的肽组学专属工作流程。
核心功能
- 在工作流中整合了 de novo sequencing, 序列数据库搜索和同源搜索
- 为多肽的de novo测序结果提供全局的FDR评估
- 深度学习模型训练与大型HLA肽数据集的保留时间,碎片离子,离子迁移预测
以前,对于多肽的从头测序结果,无法进行FDR评估,因此通常用户会选择一个ALC的打分阈值来过滤低质量的结果。而PEAKS创新性地通过将de novo序列添加到蛋白质序列数据库并执行第二轮数据库搜索,估计FDR用于de novo肽以提高识别的准确性。
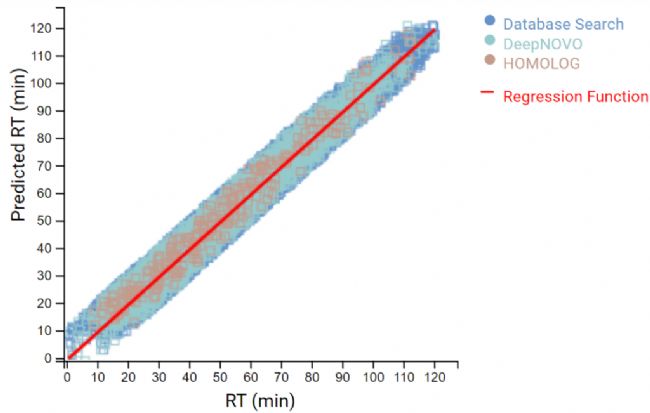
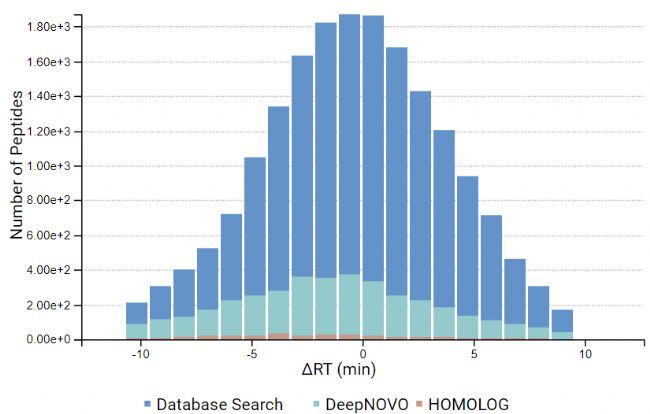
DeepNovo 多肽组工作流提供了多肽的完整鉴定列表,可以根据它们的鉴定方法进行筛选:数据库搜索,同源性搜索(具有突变的肽),或来自于从头测序的结果。结果总结页面中,用户通过统计图评估来自不同鉴定来源肽的保留时间与预测保留时间的相关性。
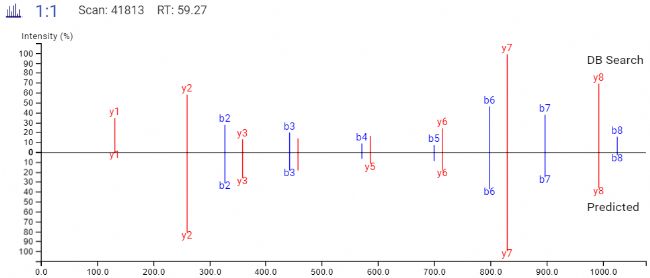
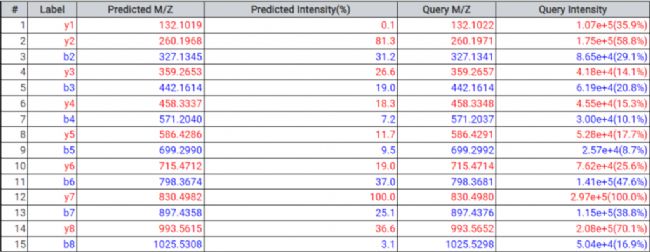
通过在结果列表中选择任意特定的肽,可以将每个谱图中碎片离子的m/z和强度值与预测的谱图进行比较。对于离子淌度数据,也预测了每个多肽的碰撞横截面积(CCS)的值。
通过在结果列表中选择任意特定的肽,可以将每个谱图中碎片离子的m/z和强度值与预测的谱图进行比较。对于离子淌度数据,也预测了每个多肽的碰撞横截面积(CCS)的值。
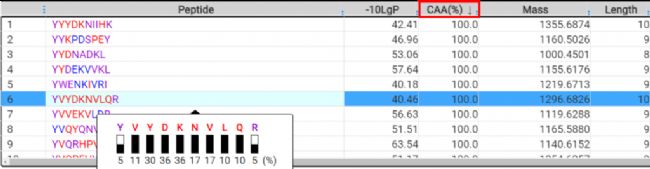
DeepNovo Peptidome工作流中添加了一种新的结果过滤方法,称为可信氨基酸百分比(confident amino acid percentage, CAA%),允许用户评估肽谱中,支持这条肽段匹配的碎片离子的信号强度值高于指定阈值的氨基酸百分比。这增加了肽序列验证的可信度。最终的多肽列表可以直接输出,用于下游分析,如结合亲和力和免疫原性预测。
参考文献
1. Tran NH, Qiao R, Xin L, Chen X, Liu C, Zhang X, Shan B, Ghodsi A, Li M. Deep learning enables de novo peptide sequencing from data-independent-acquisition mass spectrometry. Nature Methods. 16(1), 63-66. 20/12/2018.
2. Tran NH, Zhang X, Xin L, Shan B, Li M. De novo peptide sequencing by deep learning. Proceedings of the National Academy of Sciences of the United States of America. 114(29). 18/7/2017.
3. Xin, L., Qiao, R., Chen, X. et al. A streamlined platform for analyzing tera-scale DDA and DIA mass spectrometry data enables highly sensitive immunopeptidomics. Nat Commun 13, 3108 (2022).https://doi.org/10.1038/s41467-022-30867-7

2. Tran NH, Zhang X, Xin L, Shan B, Li M. De novo peptide sequencing by deep learning. Proceedings of the National Academy of Sciences of the United States of America. 114(29). 18/7/2017.
3. Xin, L., Qiao, R., Chen, X. et al. A streamlined platform for analyzing tera-scale DDA and DIA mass spectrometry data enables highly sensitive immunopeptidomics. Nat Commun 13, 3108 (2022).https://doi.org/10.1038/s41467-022-30867-7
如果您想深入了解更多关于肽组学专属工作流程DeepNovo Peptidome介绍和应用,欢迎扫描下方二维码关注我们!






