JuLI™系列实时活细胞成像分析系统助力体外胚胎模型构建
《Nature Methods》期刊在2023年12月6日发表了一篇名为《Method of the Year 2023: methods for modeling development》[1] 的文章,文中将胚胎建模作为2023年的年度最佳方法。
那么这篇文章主要内容是什么呢?以下便是对这篇文章的内容解析。
在相关领域方法开发的支持下,体外胚胎模型的研究彻底改变了我们对胚胎发生发育的理解。
发育过程始于单个细胞,然后以多细胞形式发展,并经历一轮又一轮的细胞命运决定机制和组织形态模式,最终形成一个复杂的机体结构。长期以来,由于技术和伦理方面的限制,研究人员一直无法掌握在妊娠期间控制受精卵发育成胎儿的复杂机制。然而,在过去的几年里,方法开发取得了巨大的进步,揭示了胚胎发育的这些“黑匣子”事件。
图片来源:Berna Sozen,Zernicka-Goetz 实验室
该领域的开创性方法已经成功在体外重现了胚胎发育的关键阶段,从而重塑了我们对发育的理解。例如,2021年的两篇具有里程碑意义的论文[2,3] 报道了人类囊胚的体外模型,这是胚胎着床前的早期发育阶段。今年早些时候,有研究小组[4] 报告了一个人原肠前胚胎模型,该模型概括了羊膜和卵黄囊腔的形成、早期神经发育和器官发生等关键事件。
通过结合胚胎外组织来研究植入后发生的事件,进一步完善这些模型。例如,2023年发表的两项研究报告称[5,6],人类干细胞衍生的胚胎模型可以在体内重现胚胎植入后14天的胚胎事件。
这些用于模拟人类发生发展的方法紧随技术进步和小鼠研究中首次报道的成就之后。两篇开创性论文[7,8] 描述了源自小鼠干细胞的体外模型,该模型可以在受精后长达8.5天的子宫内重现天然小鼠胚胎。这些胚胎发育到器官发生阶段,与胚外组织共同发育。该模型在形态学和转录组学分析上与天然胚胎相似,且其复杂性导致了更真实的器官和组织发育。然而,虽然可以从动物模型的发育过程中得出结论,但在这种情况下,人类胚胎发育的物种特异性特征很容易被遗漏。
由于这些复杂的模型用于深刻理解胚胎发育的机制,我们选择了体外胚胎模型构建的方法作为2023年的年度方法。
在本期特刊中,Magdalena Zernicka-Goetz[9] 深入探讨了报道的新胚胎模型。这些模型可以帮助研究人员研究形态发生背后的分子机制和组织形态模式背后的信号线索。Zernicka-Goetz提示研究人员,虽然这些胚胎模型带来了新的见解,但它们远非是对体内胚胎的完美再现。因此,当务之急是要意识到系统的局限性以及它如何影响得出的研究推论。
体外模型的真实度必须始终针对于体内来源的组织进行验证。就胚胎组织而言,由于用于研究的胚胎有限以及围绕人类胚胎操作的伦理问题,这构成了一个特殊的挑战。Muzlifah Haniffa[10] 及其同事讨论了单细胞多组学技术的出现以及随后的发育细胞图谱如何成为测试胚胎模型有效性的重要基准。
除了细胞图谱的发展之外,单细胞技术也导致了复杂的谱系追踪和轨迹分析方法的出现。尽管这些方法尚未广泛用于人类胚胎模型,但它们在绘制细胞命运事件图谱方面发挥了重要作用,例如在人脑类器官[11] 和斑马鱼胚胎[12] 中。在一篇综述中,Bushra Raj[13] 描述了该领域方法论的新进展及其在研究发展快照方面的潜力。
几十年来对哺乳动物胚胎体外培养方法的研究支持了先进的胚胎模型。Hongmei Wang及其同事在他们的评论中探讨了这一点[14]。他们写道,为了优化体外胚胎的培养条件,仍然需要开发模仿胚胎生理微环境的生物材料,以及研究细胞在胚胎发生阶段所经历的机械环境的方法。
这一观点在另一篇综述中得到了Idse Heemskerk[15] 及其同事的赞同,该综述讨论了最近的方法,这些方法现在允许研究人员绘制发育中的胚胎内的力量。综述探讨了胚胎模型如何有可能阐明组织力学、模式和形态发生之间的相互作用。与此相关的是,Hervé Turlier[16] 及其同事在同一期杂志上发表了一篇研究论文,报告了一种从小鼠和无序胚胎的3D荧光图像中进行力推断的方法。由Noah Mitchell和Dillon Cislo撰写的第二篇研究论文介绍了TubULAR16[17],这是一种用于分析动态组织在形态发生过程中变形的组织制图方法。
计算胚胎学是研究胚胎发育的一种新兴方法。研究人员探索如何通过计算生成和干扰从实验数据构建的虚拟胚胎。Patrick müller[18] 团队的一篇研究文章描述了一种基于深度学习的方法,可以对不同时间点的胚胎之间的相似性进行分级。在《Nature Methods》早期的一期文章中,同一个团队发表了EmbryoNet[19],可以分析斑马鱼胚胎表型并将它们与主要信号通路联系起来。我们的新闻特辑[20]还采访了这一领域的研究人员,他们分享了关于数字胚胎及其未来的希望和挑战。
在讨论胚胎研究的进展时,不得不考虑人类胚胎模型的伦理要求。伦理学家Nienke de Graeff、Lien de Proost和Megan Munsie在他们的综述中[21] 解释了胚胎模型带来了新的伦理问题。他们讨论了胚胎模型是否应该遵循与类器官相同的指导方针,或者它们的发育潜力是否能赋予了它们新的地位。
胚胎模型,特别是人体系统,是一个令人兴奋的新领域,得到了单细胞组学、生物材料和机械生物学领域并行方法开发的支持。我们必须就这些方法的局限性以及体外人类胚胎模型的伦理风险进行公开透明的探讨。同时,我们希望您能和我们一起努力,因为我们认识到这些方法不仅在揭示胚胎发生的细节方面具有潜力,而且在建模发育和妊娠相关疾病建模方面也具有潜力。
那么有什么样的工具可以有助于体外胚胎建模方法呢?
奎克泰生物的JuLI™系列实时活细胞成像分析系统能够为体外胚胎建模实验提供所需要的工具。
无需复杂人工操作,省时省力;无需频繁取出样本影响稳定生长环境;可全面呈现胚胎培养情况;可获取大量数据进行分析。
JuLI™ Stage活细胞成像分析系统能够放置于培养箱内,具有全自动X-Y-Z轴、三色荧光、自动/手动对焦、Z-Stack、图像拼接等功能。
可以对胚胎模型进行实时观察,拍摄记录生长周期的全过程,在保持胚胎生长环境稳定的情况下,同时对胚胎模型拍照,形成视频,提供体外胚胎培养实验的量化结果。

JuLI™ Stage拍摄胚胎实验流程:
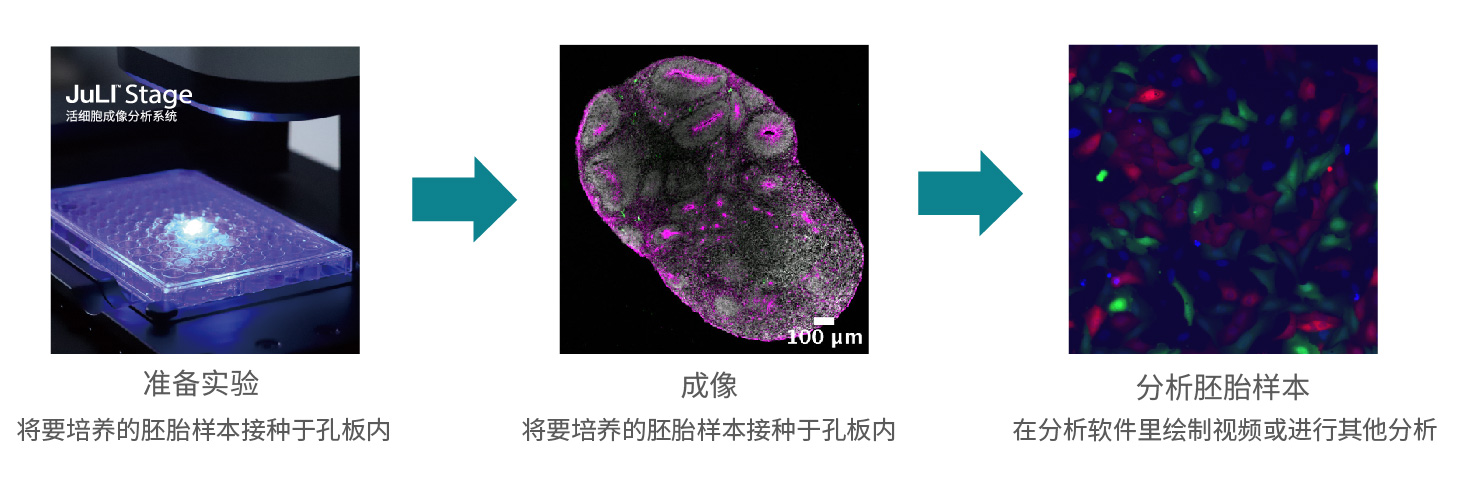
自动化的活细胞成像分析系统可以捕获最佳质量的胚胎模型图像,将大大提高胚胎体外建模以及相关实验的成功率。
我们希望您喜欢今年的年度方法,奎克泰生物祝您在2024新的一年研究顺利、取得佳绩,度过幸福溢满的一年!
参考文献:
[1] ethod of the Year 2023: methods for modeling development. Nat Methods. 2023 Dec;20(12):1831-1832.
[2] Liu X, Tan JP, Schröder J, Aberkane A, Ouyang JF, Mohenska M, Lim SM, Sun YBY, Chen J, Sun G, Zhou Y, Poppe D, Lister R, Clark AT, Rackham OJL, Zenker J, Polo JM. Modelling human blastocysts by reprogramming fibroblasts into iBlastoids. Nature. 2021 Mar;591(7851):627-632.
[3] Yu L, Wei Y, Duan J, Schmitz DA, Sakurai M, Wang L, Wang K, Zhao S, Hon GC, Wu J. Blastocyst-like structures generated from human pluripotent stem cells. Nature. 2021 Mar;591(7851):620-626.
[4] Liu L, Oura S, Markham Z, Hamilton JN, Skory RM, Li L, Sakurai M, Wang L, Pinzon-Arteaga CA, Plachta N, Hon GC, Wu J. Modeling post-implantation stages of human development into early organogenesis with stem-cell-derived peri- gastruloids. Cell. 2023 Aug 31;186(18):3776-3792.e16.
[5] Oldak B, Wildschutz E, Bondarenko V, Comar MY, Zhao C, Aguilera-Castrejon A, Tarazi S, Viukov S, Pham TXA, Ashouokhi S, Lokshtanov D, Roncato F, Ariel E, Rose M, Livnat N, Shani T, Joubran C, Cohen R, Addadi Y, Chemla M, Kedmi M, Keren-Shaul H, Pasque V, Petropoulos S, Lanner F, Novershtern N, Hanna JH. Complete human day 14 post-implantation embryo models from naive ES cells. Nature. 2023 Oct;622(7983):562-573.
[6] Weatherbee BAT, Gantner CW, Iwamoto-Stohl LK, Daza RM, Hamazaki N, Shendure J, Zernicka-Goetz M. Pluripotent stem cell-derived model of the post-implantation human embryo. Nature. 2023 Oct;622(7983):584-593.
[7] Tarazi S, Aguilera-Castrejon A, Joubran C, Ghanem N, Ashouokhi S, Roncato F, Wildschutz E, Haddad M, Oldak B, Gomez-Cesar E, Livnat N, Viukov S, Lokshtanov D, Naveh-Tassa S, Rose M, Hanna S, Raanan C, Brenner O, Kedmi M, Keren-Shaul H, Lapidot T, Maza I, Novershtern N, Hanna JH. Post-gastrulation synthetic embryos generated ex utero from mouse naive ESCs. Cell. 2022 Sep 1;185(18):3290-3306.e25.
[8] Amadei G, Handford CE, Qiu C, De Jonghe J, Greenfeld H, Tran M, Martin BK, Chen DY, Aguilera-Castrejon A, Hanna JH, Elowitz MB, Hollfelder F, Shendure J, Glover DM, Zernicka-Goetz M. Embryo model completes gastrulation to neurulation and organogenesis. Nature. 2022 Oct;610(7930):143-153.
[9] Zernicka-Goetz M. The evolution of embryo models. Nat Methods. 2023 Dec;20(12):1844-1848.
[10] Haniffa M, Maartens A, Teichmann SA. How developmental cell atlases inform stem cell embryo models. Nat Methods. 2023 Dec;20(12):1849-1851.
[11] He Z, Maynard A, Jain A, Gerber T, Petri R, Lin HC, Santel M, Ly K, Dupré JS, Sidow L, Sanchis Calleja F, Jansen SMJ, Riesenberg S, Camp JG, Treutlein B. Lineage recording in human cerebral organoids. Nat Methods. 2022 Jan;19(1):90-99.
[12] Lange, M. et al. Preprint at bioRxiv https://doi.org/10.1101/2023.03.06.531398 (2023)
[13] Raj B. Tracing developmental lineages. Nat Methods. 2023 Dec;20(12):1852-1854.
[14] Wu X, Zhai J, Li Q, Wang H. The in vitro culture of mammalian embryos. Nat Methods. 2023 Dec;20(12):1855-1858.
[15] Liu ZY, Trivedi V, Heemskerk I. Mapping morphogenesis and mechanics in embryo models. Nat Methods. 2023 Dec;20(12):1859-1862.
[16] Ichbiah S, Delbary F, McDougall A, Dumollard R, Turlier H. Embryo mechanics cartography: inference of 3D force atlases from fluorescence microscopy.
Nat Methods. 2023 Dec;20(12):1989-1999.
[17] Mitchell NP, Cislo DJ. TubULAR: tracking in toto deformations of dynamic tissues via constrained maps. Nat Methods. 2023 Dec;20(12):1980-1988.
[18] Toulany N, Morales-Navarrete H, Čapek D, Grathwohl J, Ünalan M, Müller P. Uncovering developmental time and tempo using deep learning. Nat Methods. 2023 Dec;20(12):2000-2010.
[19] Čapek D, Safroshkin M, Morales-Navarrete H, Toulany N, Arutyunov G, Kurzbach A, Bihler J, Hagauer J, Kick S, Jones F, Jordan B, Müller P. EmbryoNet: using deep learning to link embryonic phenotypes to signaling pathways. Nat Methods. 2023 Jun;20(6):815-823.
[20] Marx V. How to build a virtual embryo. Nat Methods. 2023 Dec;20(12):1838-1843.
[21]de Graeff N, De Proost L, Munsie M. 'Ceci n'est pas un embryon?' The ethics of human embryo model research. Nat Methods. 2023 Dec;20(12):1863-1867.





