|
目标区域测序(Target Region Sequencing, TRS)是根据感兴趣的基因组区域设计特异性探针,与基因组DNA进行液相杂交,将目标基因组区域的DNA片段进行富集后再利用第二代测序技术进行测序的研究策略。
|
||||||||||||||||||
| [发表评论] [本类其他服务] [本类其他服务商] | ||||||||||||||||||
| 服务商: 深圳华大基因科技服务有限公司 | 查看该公司所有服务 >> |
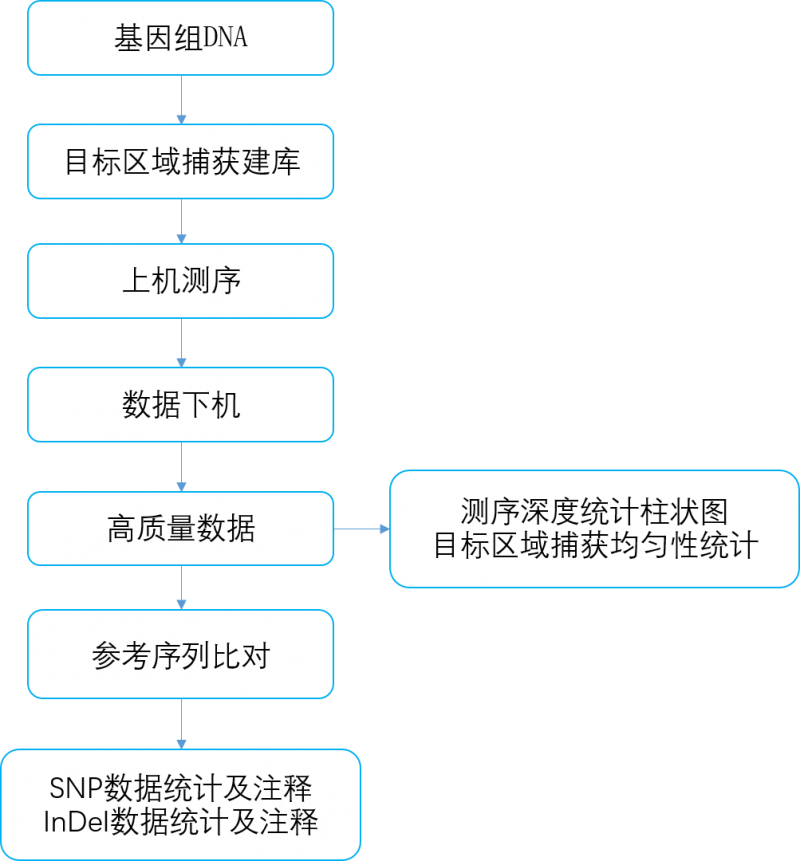
图1 技术流程
华大可提供以下两种目标区域捕获探针:Agilent SureSelect Target Enrichment System及NimbleGen SeqCap EZ Choice。
1、Agilent捕获系统
Agilent SureSelect Target Enrichment System液相捕获,是基于120mer的RNA寡核苷酸探针或者叫“baits”。Baits上连接的生物素,可以被链霉亲和素标记的磁珠吸附。打断后的基因组片段,与baits进行杂交,捕获目标片段。利用磁珠吸附出带有baits的DNA片段后,进行磁珠洗脱、RNA探针降解,最终获得目标区域DNA片段。
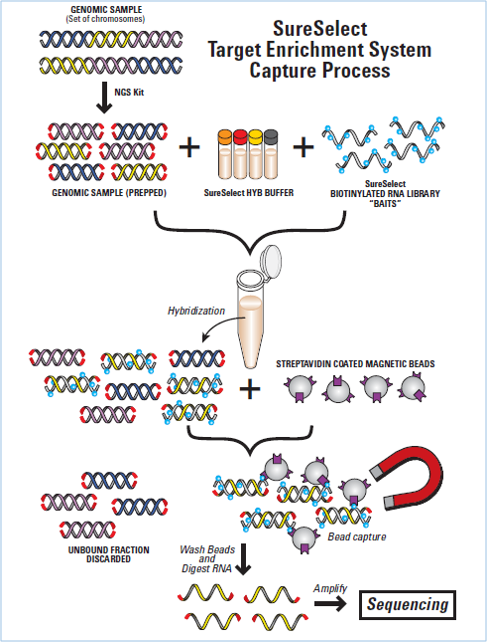
图2 Agilent SureSelect捕获流程
2、NimbleGen捕获系统
与Agilent的捕获原理类似,NimbleGen采用DNA探针,以高密度探针著称,因此价格也相对较高。如下图所示,NimbleGen利用高密度的50-105mer的DNA探针来覆盖目标区域。
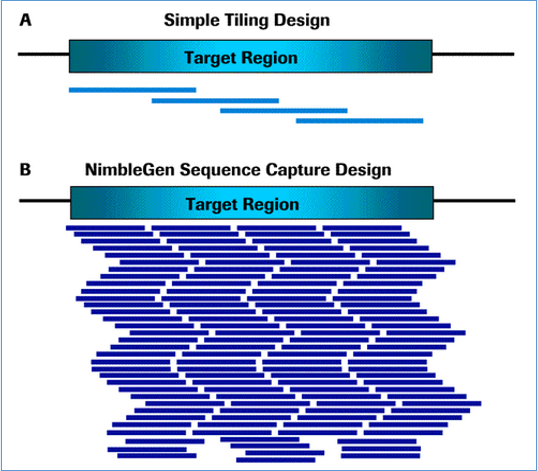
图3 NimbleGen探针设计示意图
定制化芯片型号
|
芯片名称 |
Reactions/kit |
芯片大小 |
|
Agilent Sureselect XT Custom Kits |
16, 96 |
1~499Kb,0.5~2.9Mb,3~5.9Mb,6Mb~11.9Mb,12Mb~24Mb |
|
NimbleGen SeqCap EZ Choice Kits |
12, 24, 48, 96, 384, 960 |
100kb-7Mb |
|
NimbleGen SeqCap EZ Choice XL Kits |
12, 24, 48, 96, 384, 960 |
7Mb-200Mb |
标准信息分析
1. 去除接头污染和低质量数据
2. 数据通过BWA与UCSC hg19数据库进行比对
3. 数据产量统计分析、测序深度分析、覆盖度均一性分析
4. SNP变异信息检测(SAMtools、SOAPsnp、GATK)
5. SNP的RefGene注释
6. SNP数据库分析(与dbSNP、千人基因组数据、ESP外显子组数据库以及炎黄基因组(仅亚太地区)数据进行数据库注释分析)
7. 单样品SNP保守性预测、致病性分析(仅针对人类样本,软件:SIFT、Polyphen-2、Phylop、GERP scores、Mutation assessor、Condel、FATHMM)
8. SNP在各基因功能元件上的分布统计
9. InDel变异信息检测(SAMtools、GATK)
10. InDel的RefGene注释
11. InDel数据库分析(与dbSNP 、千人基因组数据、ESP外显子组数据库、炎黄基因组(仅亚太地区)进行数据库注释分析)
12. InDel在各基因功能元件上的分布统计
注:SIFT、Polyphen-2、Phylop、GERP scores、Mutation assessor、Condel、FATHMM这几个数据库的分析仅针对人类样本。



