超长(Ultra-long)DNA核酸片段回收仪的工作原理
随着基因组学的发展,短读长测序技术在解析复杂基因组区域、检测结构变异以及表观遗传学研究等方面,其序列拼接常常导致缺失和错误,从而激发了各种长读长测序策略的发展,也让研究者更加期望可以一次完整获取目标全长序列,从而获取更准确、更全面的序列信息。来自美国Sage Science公司研发生产的HLS超长(Ultra-long)DNA片段回收系统, 整合了脉冲场电泳,直接从提取的基因组样品中完成DNA超大片段的分选回收,或者从细胞样本中提取、纯化DNA并完成DNA超大片段的分选回收,用于制备百kb以上的文库,结合长片段测序可以进一步提高read长度,提高测序质量和效率,挖掘新的序列信息。

超长核酸片段回收仪(50kb-2Mb DNA片段)
HLS可回收50kb-2Mb DNA片段,用于超长DNA测序文库制备,更好的服务于基因组组装和结构变异测序的研究。此外,Sage Science公司联合美国国家基因组研究所共同开发出独有的长片段DNA靶向获取技术:HLS-CATCH技术,该技术使用Cas9基因编辑技术从基因组上直接靶向获取50-400kb的完整大片段DNA序列,用于下游序列分析。HLS还可以应用于染色体外DNA的回收,比如线粒体DNA回收等。
- 1.超长片段回收
HLS超长(Ultra-long)DNA片段回收功能,广泛应用于三代测序领域。例如,ultra-long Nanopore library制备过程中,经过HLS自动化片段筛选后,可以将短片段DNA过滤掉,最大限度保留长片段DNA,充分发挥Nanopore测序仪长读长测序优势,获取更全面的基因组信息。
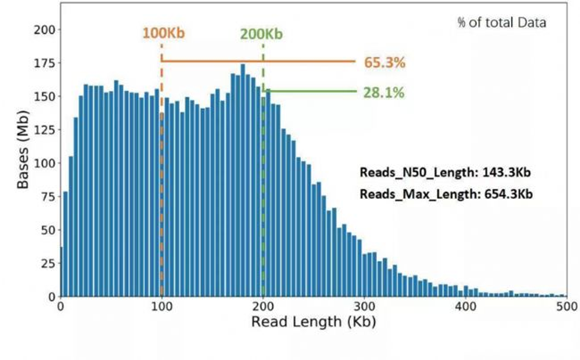
经过HLS片段筛选,reads N50 length可达143.3kb
(数据来源:武汉未来组Nanopore测序)
(数据来源:武汉未来组Nanopore测序)
HLS用于制备ultra-long Nanopore library文献:
| 序号 | 文献 | 日期 | 物种 | 测序平台 |
| 1 | Huang, Zhen, et al. "Evolutionary analysis of a complete chicken genome." Proceedings of the National Academy of Sciences 120.8 (2023): e2216641120. | 2023年 | 鸡 | Oxford Nanopore Technologies |
| 2 | Guo, Xing, et al. "The genome of Acorus deciphers insights into early monocot evolution." Nature Communications 14.1 (2023): 3662. | 2023年 | 菖蒲 | Oxford Nanopore Technologies |
| 3 | Wang, Qing, et al. "Draft genome of the oriental garden lizard (Calotes versicolor)." Frontiers in Genetics 14 (2023): 1091544. | 2023年 | 蜥蜴 | Oxford Nanopore Technologies |
| 4 | Morita, Shinichi, et al. "The draft genome sequence of the Japanese rhinoceros beetle Trypoxylus dichotomu s septentrionalis towards an understanding of horn formation." Scientific Reports 13.1 (2023): 8735. | 2023年 | 昆虫 | Oxford Nanopore Technologies |
| 5 | Ma, Fengjiao, et al. "Gap-free genome assembly of anadromous Coilia nasus." Scientific Data 10.1 (2023): 360. | 2023年 | 线圈虫 | Oxford Nanopore Technologies |
| 6 | Wang, Bo, et al. "High-quality Arabidopsis thaliana genome assembly with nanopore and HiFi long reads." Genomics, proteomics & bioinformatics 20.1 (2022): 4-13. | 2022年 | 拟南芥 | Oxford Nanopore Technologies |
| 7 | Li, Baiyuan, et al. "Genomic Island-mediated horizontal transfer of the erythromycin resistance gene erm (X) among bifidobacteria." Applied and Environmental Microbiology 88.10 (2022): e00410-22. | 2022年 | 双歧杆菌 | Oxford Nanopore Technologies |
| 8 | Bao, Zhigui, et al. "Genome architecture and tetrasomic inheritance of autotetraploid potato." Molecular Plant 15.7 (2022): 1211-1226. | 2022年 | 马铃薯 | Oxford Nanopore Technologies |
| 9 | Buitrago, Diana, et al. "Impact of DNA methylation on 3D genome structure." Nature Communications 12.1 (2021): 3243. | 2021年 | 酵母 | Oxford Nanopore Technologies |
- 2.HLS-CATCH技术

视频:HLS-CATCH技术原理
(请点击图片,观察视频链接)
HLS-CATCH技术文献
(请点击图片,观察视频链接)
| 序号 | 文献 | 日期 | 测序平台 |
| 1 | Mikhaylov, Veronika, et al. "Targeted Phasing of 2-200 Kilobase DNA Fragments with a Short-Read Sequencer and a Single-Tube Linked-Read Library Method." bioRxiv (2023): 2023-03. | 2023年 | Illumina |
| 2 | Vogelaar, Ingrid P., et al. "Large cancer pedigree involving multiple cancer genes including likely digenic MSH2 and MSH6 lynch syndrome (LS) and an instance of recombinational rescue from LS." Cancers 15.1 (2022): 228. | 2022年 | Illumina |
| 3 | Walsh, Tom, et al. "CRISPR–Cas9/long-read sequencing approach to identify cryptic mutations in BRCA1 and other tumour suppressor genes." Journal of medical genetics 58.12 (2021): 850-852. | 2021年 | Pacific Biosciences |
| 4 | Zhou, Bo, et al. "Complete and haplotype-specific sequence assembly of segmental duplication-mediated genome rearrangements using targeted CRISPR-targeted ultra-long read sequencing (CTLR-Seq)." bioRxiv (2020): 2020-10. | 2020年 | Oxford Nanopore Technologies |
| 5 | Shin, GiWon, et al. "Targeted short read sequencing and assembly of re-arrangements and candidate gene loci provide megabase diplotypes." Nucleic acids research 47.19 (2019): e115-e115. | 2019年 | Illumina |
- 3.线粒体DNA富集

线粒体DNA富集原理图
Sage Science是全球领先的全自动核酸电泳片段回收仪的生产厂家,拥有美国技术专利,各个型号产品可以高效完成不同长度范围DNA片段的精准回收,广泛用于二代测序、三代测序以及多种分子生物学相关领域。
环亚生物科技有限公司作为美国Sage Science公司在中国区的独家代理商,自2011年以来将Sage Science的产品线引入国内,一直为国内用户提供专业的全自动核酸片段回收系统的安装测试、应用培训,技术支持与售后维护工作,赢得客户的一致好评与信任。环亚生物科技有限公司将一如既往的支持越来越多的Sage Science用户。
▼更多 HLS 详情,请联系我们▼
环亚生物科技有限公司
地址:上海市闵行区友东路358号闵欣大厦1号楼701/703/705室
电话:021-54583565
邮箱:info@apgbio.com
网址:www.apgbio.com
▼更多精彩内容,请扫码关注我们▼







