利用X射线显微断层扫描技术对蜥蜴脑部进行虚拟组织学研究操作流程
Zhou, Tunhe, Yulia Dragunova, and Zegni Triki. “Brain Virtual Histology of a Lizard Species (Podarcis Bocagei) Using X-Ray Micro-Tomography and Deep-Learning Segmentation.” bioRxiv, July 6, 2024. https://doi.org/10.1101/2024.07.05.602071.
本文旨在展示如何利用X射线显微断层扫描技术(microCT)和深度学习分割,对蜥蜴(Podarcis bocagei)的脑部进行虚拟组织学研究,并提供一个详细的操作流程。
01 研究背景
02 Dragonfly软件的使用
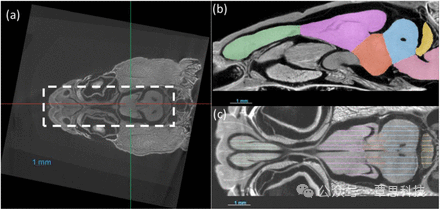 (a) The images are aligned and cropped to keep the brain for fast segmentation. (b,c) Demonstration of manual segmentation.
(a) The images are aligned and cropped to keep the brain for fast segmentation. (b,c) Demonstration of manual segmentation.
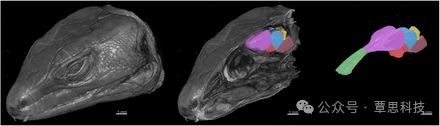
3D rendering of the X-ray microCT images showing the external and internal of the head, and the main parts of the brain. The brain regions are: the olfactory bulb (green), telencephalon (purple), diencephalon (red), midbrain (blue), cerebellum (yellow) and brainstem (pink).
03 研究方法
1. 样本准备: 对蜥蜴头部样本进行磷钨酸(PTA)染色,增强组织对比度。
2. X射线microCT扫描: 使用蔡司Xradia Versa 520扫描仪对样本进行扫描,获取高分辨率的3D图像。
3. 半手动分割训练集: 在Dragonfly中手动分割部分切片,然后使用Biomedisa软件进行随机游走插值,最后手动检查和修正。
4. 深度学习分割: 比较了Biomedisa和AIMOS两种深度学习算法,使用训练好的模型对剩余数据进行自动分割。
5. 分割评估: 使用平均dice score(DSC)和脑区体积平均相对误差(RE)评估分割精度。
04研究结果
结论
Dragonfly软件在本研究中主要用于图像预处理(对齐和裁剪)、手动分割训练集的创建,以及3D模型重建和可视化等方面,为后续的深度学习分割和分析提供了基础。
本文旨在展示如何利用X射线显微断层扫描技术(microCT)和深度学习分割,对蜥蜴(Podarcis bocagei)的脑部进行虚拟组织学研究,并提供一个详细的操作流程。
01 研究背景
- 传统的组织学方法耗时且对组织具有破坏性,而microCT可以提供高分辨率的3D图像,实现任意方向的虚拟组织学切片,减少了对样本的破坏和所需动物数量。
- 虽然磁共振成像(MRI)也被用于脑部研究,但microCT在骨骼和肌肉组织方面具有更高的对比度和分辨率,能提供更全面的头部解剖细节。
- 深度学习分割在医学影像领域发展迅速,但野生动物研究通常样本量较小。本研究旨在证明深度学习分割在小样本量下的有效性。
02 Dragonfly软件的使用
- 图像对齐与裁剪: 由于样本放置方向不一致,作者使用Dragonfly软件对图像进行对齐和裁剪,确保脑部区域一致,方便后续分析和深度学习训练。
- 手动分割训练集: 作者在Dragonfly中手动分割了约20-30个切片,作为深度学习模型的训练集。
- 3D模型重建与可视化: 作者使用Dragonfly将microCT图像重建为3D模型,并对分割后的脑区进行可视化展示。


3D rendering of the X-ray microCT images showing the external and internal of the head, and the main parts of the brain. The brain regions are: the olfactory bulb (green), telencephalon (purple), diencephalon (red), midbrain (blue), cerebellum (yellow) and brainstem (pink).
03 研究方法
1. 样本准备: 对蜥蜴头部样本进行磷钨酸(PTA)染色,增强组织对比度。
2. X射线microCT扫描: 使用蔡司Xradia Versa 520扫描仪对样本进行扫描,获取高分辨率的3D图像。
3. 半手动分割训练集: 在Dragonfly中手动分割部分切片,然后使用Biomedisa软件进行随机游走插值,最后手动检查和修正。
4. 深度学习分割: 比较了Biomedisa和AIMOS两种深度学习算法,使用训练好的模型对剩余数据进行自动分割。
5. 分割评估: 使用平均dice score(DSC)和脑区体积平均相对误差(RE)评估分割精度。
04研究结果
- 成功分割出29个样本的六个主要脑区:嗅球、端脑、间脑、中脑、小脑和脑干。
- 两种深度学习算法均只需5个训练集即可达到较高的分割精度(DSC>0.94),且与手动分割的体积测量差异约为4%。
- 不同切片方向(冠状面、矢状面和水平面)对分割精度无显著影响。
结论
- 本研究提供了一种高效、准确的蜥蜴脑部3D成像和体积测量方法。
- microCT结合深度学习分割,特别适用于样本量较小的野生动物研究。
- 该方法在生态学、进化生物学等领域具有广泛应用前景,并益于对头部其他解剖细节感兴趣的研究,以及计算机科学家开发更高效的算法。
Dragonfly软件在本研究中主要用于图像预处理(对齐和裁剪)、手动分割训练集的创建,以及3D模型重建和可视化等方面,为后续的深度学习分割和分析提供了基础。





