InnoScan在环境蓝细菌高通量检测微阵列芯片的应用
InnoScan文献快讯—
环境蓝细菌高通量检测微阵列芯片

摘要:环境蓝细菌因其在生态系统中的关键作用而备受关注。 中国研究团队近期开发了一种名为 CyanoStrainChip 的新型DNA 微阵列芯片,可对环境蓝细菌进行菌株水平的全面分析。 通过 43,666个全基因组、菌株特异性探针涵盖几乎所有蓝细菌主要类群。 使用CyanoStrainChip无需培养蓝细菌,与传统方法相比成本更低,计算要求更低,定量效果更好。

蓝细菌代表一个广泛而普遍存在的光合自养原核生物门,以其非凡的遗传多样性而闻名。这些微生物存在于几乎所有环境中,并在生态系统中发挥关键作用,不仅是地球大气氧气的重要贡献者,还是次生代谢产物的多产者。在某些情况下,蓝细菌会导致有害的水华,释放毒素进入淡水和海洋环境,对生态系统结构和功能,以及用于娱乐、饮用水、渔业和人类健康的水质构成严重威胁。事实上,在各种生态系统中对蓝细菌多样性和丰度进行分析将对生态研究和有害藻华监测、评估和管理至关重要。
然而,当前研究普遍忽视了蓝细菌种内多样性,导致在种或以上的概括性结论,并未考虑菌株差异的可能性。许多属于同一蓝细菌属(或种)的多个分离株比较研究,如 Raphidiopsis、Planktothrix 和 Microcystis,证明它们在形态、生理、毒素和遗传学方面存在相当大的种内多样性。但是目前环境蓝细菌的高通量检测方法很难达到菌株水平。基于聚合酶链反应 (PCR) 的靶向标记基因DNA 测序是克服蓝细菌多样性评估中表型表征局限性,最广泛使用的技术。早在1997年,就开发出了一组用于特异性扩增蓝细菌16S rRNA基因片段的寡核苷酸引物。然而,这些基于PCR的测序方法无法在物种或以下进行鉴定,因为DNA片段只有几百个核苷酸长,其分辨率受到限制。过去十年来,对环境样本核酸进行直接宏基因组测序已成为探索复杂环境微生物群落组成的有力工具。虽然通过生物信息学工具(如 inStrain 和 PStrain)可成功识别群落中的菌株,但仍然存在一定的局限性。鉴于环境样品的高复杂性,通常需要足够的序列深度来提高灵敏度和特异性,这也意味着巨大的测序和计算成本,这对许多研究人员来说仍然不可负担。此外,技术重现性低,并且在宏基因组分析中准确量化密切相关菌株的相对丰度存在困难,可能会引入显著偏差。因此,需要一种新方法来解决现有方法的这些缺点。
DNA微阵列最初由数万个DNA片段排列在载玻璃片上,以进行基因表达分析。从那时起,各种类型的DNA微阵列已被开发用于不同环境中的微生物检测,包括功能和分类分析。与基于测序的方法相比,微阵列具有几个优势,包括高重复性、可靠的定量、相对可负担的成本和易于使用。鉴于高密度微阵列技术的成熟和蓝细菌基因组数据的快速积累,近期中国一研究团队开发了 CyanoStrainChip,这是一种DNA微阵列芯片,用于环境蓝细菌的高通量和高分辨率分类鉴定。该团队设计了43,666个全基因组菌株特异性探针,用于靶向1277株蓝细菌。通过体外模拟群落和野外样品检查该芯片的实用性和局限性。结果表明,该微阵列芯片能够在复杂环境中准确地在菌种水平检测蓝细菌。
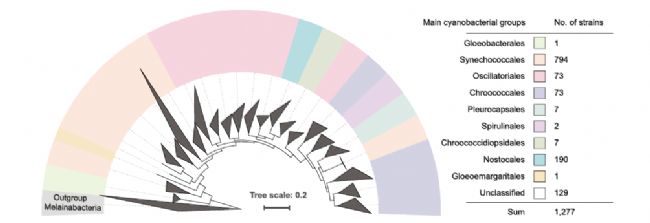
CyanoStrainChip目标蓝细菌菌株按主要类群呈现。蓝细菌主要群体的分支关系,基于先前研究的系统发育分析,利用了834个蓝细菌特异性通用单拷贝同源基因基准。
该芯片设计,一共使用了约2500个蓝细菌基因组来设计特异性探针,其中包括163个基因组,这些基因组在该研究团队之前的出版物中已测序,以及约2300个来自NCBI GenBank数据库的基因组。为了减少数据集的冗余,采用FastANI来生成整个基因组的相似性计算指标,并将所有基因组在99%相似性阈值下聚类成菌株。对于每个菌株簇,保留一个代表性基因组进行下游分析。总共,有1277个基因组保留下来,包括458个纯培养基因组、556个单一扩增基因组和263个宏基因组组装基因组。
采用基于k-mer的方法来发现菌株特异性寡核苷酸,根据寡核苷酸的各种性质(包括探针杂交自由能、熔解温度、探针次级结构、特异性和复杂性)对候选探针进行过滤。对于每个菌株,如果剩余探针足够多,则选择30-40个特异性探针;否则,保留所有剩余探针。选择的探针被提交到Agilent eArray 5.0程序进行微阵列定制(客制CGH,8×64K平台)。
CyanoStrainChip数据的预处理。在InnoScan 900扫描仪(该型号现已更新为InnoScan 910) 上扫描的微阵列tiff图像通过Agilent Feature Extraction(AFE)软件处理,以生成样品点原始强度和一系列统计指标。在进行下游分析之前,采取几个微阵列数据预处理步骤来抵消系统性变化并过滤假阳性。

文献原文链接: https://doi.org/10.1021/acs.est.3c11096
环境蓝细菌高通量检测微阵列芯片

摘要:环境蓝细菌因其在生态系统中的关键作用而备受关注。 中国研究团队近期开发了一种名为 CyanoStrainChip 的新型DNA 微阵列芯片,可对环境蓝细菌进行菌株水平的全面分析。 通过 43,666个全基因组、菌株特异性探针涵盖几乎所有蓝细菌主要类群。 使用CyanoStrainChip无需培养蓝细菌,与传统方法相比成本更低,计算要求更低,定量效果更好。

蓝细菌代表一个广泛而普遍存在的光合自养原核生物门,以其非凡的遗传多样性而闻名。这些微生物存在于几乎所有环境中,并在生态系统中发挥关键作用,不仅是地球大气氧气的重要贡献者,还是次生代谢产物的多产者。在某些情况下,蓝细菌会导致有害的水华,释放毒素进入淡水和海洋环境,对生态系统结构和功能,以及用于娱乐、饮用水、渔业和人类健康的水质构成严重威胁。事实上,在各种生态系统中对蓝细菌多样性和丰度进行分析将对生态研究和有害藻华监测、评估和管理至关重要。
然而,当前研究普遍忽视了蓝细菌种内多样性,导致在种或以上的概括性结论,并未考虑菌株差异的可能性。许多属于同一蓝细菌属(或种)的多个分离株比较研究,如 Raphidiopsis、Planktothrix 和 Microcystis,证明它们在形态、生理、毒素和遗传学方面存在相当大的种内多样性。但是目前环境蓝细菌的高通量检测方法很难达到菌株水平。基于聚合酶链反应 (PCR) 的靶向标记基因DNA 测序是克服蓝细菌多样性评估中表型表征局限性,最广泛使用的技术。早在1997年,就开发出了一组用于特异性扩增蓝细菌16S rRNA基因片段的寡核苷酸引物。然而,这些基于PCR的测序方法无法在物种或以下进行鉴定,因为DNA片段只有几百个核苷酸长,其分辨率受到限制。过去十年来,对环境样本核酸进行直接宏基因组测序已成为探索复杂环境微生物群落组成的有力工具。虽然通过生物信息学工具(如 inStrain 和 PStrain)可成功识别群落中的菌株,但仍然存在一定的局限性。鉴于环境样品的高复杂性,通常需要足够的序列深度来提高灵敏度和特异性,这也意味着巨大的测序和计算成本,这对许多研究人员来说仍然不可负担。此外,技术重现性低,并且在宏基因组分析中准确量化密切相关菌株的相对丰度存在困难,可能会引入显著偏差。因此,需要一种新方法来解决现有方法的这些缺点。
DNA微阵列最初由数万个DNA片段排列在载玻璃片上,以进行基因表达分析。从那时起,各种类型的DNA微阵列已被开发用于不同环境中的微生物检测,包括功能和分类分析。与基于测序的方法相比,微阵列具有几个优势,包括高重复性、可靠的定量、相对可负担的成本和易于使用。鉴于高密度微阵列技术的成熟和蓝细菌基因组数据的快速积累,近期中国一研究团队开发了 CyanoStrainChip,这是一种DNA微阵列芯片,用于环境蓝细菌的高通量和高分辨率分类鉴定。该团队设计了43,666个全基因组菌株特异性探针,用于靶向1277株蓝细菌。通过体外模拟群落和野外样品检查该芯片的实用性和局限性。结果表明,该微阵列芯片能够在复杂环境中准确地在菌种水平检测蓝细菌。

CyanoStrainChip目标蓝细菌菌株按主要类群呈现。蓝细菌主要群体的分支关系,基于先前研究的系统发育分析,利用了834个蓝细菌特异性通用单拷贝同源基因基准。
该芯片设计,一共使用了约2500个蓝细菌基因组来设计特异性探针,其中包括163个基因组,这些基因组在该研究团队之前的出版物中已测序,以及约2300个来自NCBI GenBank数据库的基因组。为了减少数据集的冗余,采用FastANI来生成整个基因组的相似性计算指标,并将所有基因组在99%相似性阈值下聚类成菌株。对于每个菌株簇,保留一个代表性基因组进行下游分析。总共,有1277个基因组保留下来,包括458个纯培养基因组、556个单一扩增基因组和263个宏基因组组装基因组。
采用基于k-mer的方法来发现菌株特异性寡核苷酸,根据寡核苷酸的各种性质(包括探针杂交自由能、熔解温度、探针次级结构、特异性和复杂性)对候选探针进行过滤。对于每个菌株,如果剩余探针足够多,则选择30-40个特异性探针;否则,保留所有剩余探针。选择的探针被提交到Agilent eArray 5.0程序进行微阵列定制(客制CGH,8×64K平台)。
CyanoStrainChip数据的预处理。在InnoScan 900扫描仪(该型号现已更新为InnoScan 910) 上扫描的微阵列tiff图像通过Agilent Feature Extraction(AFE)软件处理,以生成样品点原始强度和一系列统计指标。在进行下游分析之前,采取几个微阵列数据预处理步骤来抵消系统性变化并过滤假阳性。

InnoScan 910 微阵列扫描仪
文献原文链接: https://doi.org/10.1021/acs.est.3c11096





